Recherche en Psychologie
I
P01 : Laboratoire de perception et d’attention
Sevda Montakhaby Nodeh
Laboratoire de perception et d’attention
En tant que chercheur en sciences cognitives au Cognition and Attention Lab de l’Université McMaster, vous êtes à la pointe de l’exploration des subtilités du contrôle proactif dans les processus d’attention. Cette ligne de recherche est d’une grande importance, étant donné que le système sensoriel humain est submergé par une vaste gamme d’informations à chaque seconde, dépassant ce qui peut être traité de manière significative. L’essence de la recherche sur l’attention consiste à décrypter les mécanismes par lesquels l’entrée sensorielle est parcourue et gérée de manière sélective. Cela est particulièrement important pour comprendre comment les individus anticipent et s’adaptent en vue de tâches ou de stimuli à venir, un phénomène particulièrement pertinent dans des environnements regorgeant de distractions potentielles.
Dans la vie de tous les jours, les conflits attentionnels sont monnaie courante et se manifestent lorsque des informations non pertinentes par rapport à un objectif entrent en compétition avec des données pertinentes par rapport à un objectif pour la priorité attentionnelle. Un exemple de ce phénomène (que nous ne connaissons que trop bien, j’en suis sûr) est la perturbation causée par les notifications sur nos appareils mobiles, qui peuvent nous détourner de nos objectifs principaux, tels que les études ou la conduite. D’un point de vue scientifique, l’élucidation des stratégies employées par le système cognitif humain pour optimiser la sélection et le maintien d’un comportement orienté vers un objectif représente un défi formidable et irrésistible.
Votre recherche en cours est une réponse directe à ce défi. Elle examine comment le contrôle proactif influence la capacité à se concentrer sur les informations pertinentes pour la tâche tout en écartant efficacement les distractions. Cette facette de la fonctionnalité cognitive n’est pas seulement une construction théorique ; c’est le fondement même du comportement et de l’interaction humaine au quotidien.
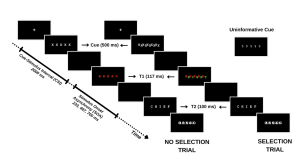
Votre étude évalue méthodiquement cette dynamique en engageant les participants dans une tâche où ils doivent identifier une séquence de mots dans des conditions variables. Le premier mot (T1) est présenté en rouge, suivi rapidement par un second mot (T2) en blanc. L’intervalle entre l’apparition de T1 et T2, connu sous le nom d’asynchronisme d’apparition du stimulus (SOA), sert de mesure critique dans votre expérience. La particularité de votre étude réside dans la façon dont vous manipulez les demandes d’attention sélective dans chaque essai, classées comme suit :
- Essais sans sélection : T1 apparaît seul, ce qui conduit généralement à une précision d’identification supérieure pour T1 et T2 en raison d’une charge cognitive réduite.
- Essais de sélection : T1 est entrecoupée d’un mot distracteur vert. Dans ces essais plus exigeants, le distracteur vert est en concurrence avec le mot cible rouge, ce qui entraîne une baisse de la précision d’identification pour T1 et T2.
En introduisant des indices informatifs et non informatifs, votre enquête sonde le rôle du contrôle proactif. Les indices informatifs donnent aux participants un aperçu du type d’épreuve à venir, ce qui leur permet de se préparer mentalement au défi imminent. À l’inverse, les indices non informatifs agissent comme des essais de contrôle et n’offrent aucune indication sur le type d’essai. L’hypothèse est que ces indices informatifs permettent aux participants d’ajuster leur attention de manière proactive en prévision d’un conflit attentionnel, ce qui pourrait améliorer les performances lors des essais de sélection avec des indices informatifs par rapport à ceux avec des indices non informatifs.
Pour une vue d’ensemble des différents types d’essais, veuillez vous référer à la figure ci-dessous. (le texte de la figure demeure toutefois en anglais seulement)
Dans le cadre de cette étude, vous vous attaquez non seulement à la question plus générale du rôle de l’effort conscient dans l’attention, mais vous contribuez également à une compréhension nuancée des processus cognitifs humains, ouvrant la voie à des applications qui vont de l’amélioration de la productivité de la vie quotidienne à l’optimisation des interfaces technologiques en vue d’une perturbation minimale de la cognition.
Mise en route : Chargement des progiciels , définition du répertoire de travail et chargement du jeu de données
Commençons par exécuter le code suivant dans RStudio pour charger les bibliothèques requises. Veillez à lire les commentaires intégrés dans le code pour comprendre ce que fait chaque ligne de code.
Remarque : les cases grisées contiennent le code R, le signe “#” indiquant un commentaire qui ne s’exécutera pas dans RStudio.
# Here we create a list called "my_packages" with all of our required libraries
my_packages <- c("tidyverse", "rstatix", "readxl", "xlsx", "emmeans", "afex",
"kableExtra", "grid", "gridExtra", "superb", "ggpubr", "lsmeans")
# Checking and extracting packages that are not already installed
not_installed <- my_packages[!(my_packages %in% installed.packages()[ , "Package"])]
# Install packages that are not already installed
if(length(not_installed)) install.packages(not_installed)
# Loading the required libraries
library(tidyverse) # for data manipulation
library(rstatix) # for statistical analyses
library(readxl) # to read excel files
library(xlsx) # to create excel files
library(kableExtra) # formatting html ANOVA tables
library(superb) # production of summary stat with adjusted error bars(Cousineau, Goulet, & Harding, 2021)
library(ggpubr) # for making plots
library(grid) # for plots
library(gridExtra) # for arranging multiple ggplots for extraction
library(lsmeans) # for pairwise comparisons
Assurez-vous d’avoir téléchargé l’ensemble de données requis (“ProactiveControlCueing.xlsx”) pour cet exercice. Définissez le répertoire de travail de votre session R actuelle dans le dossier contenant l’ensemble de données téléchargé. Vous pouvez le faire manuellement dans le studio R en cliquant sur l’onglet “Session” en haut de l’écran, puis en cliquant sur “Set Working Directory”.
Si le fichier de données téléchargé et votre session R se trouvent dans le même dossier, vous pouvez choisir de définir votre répertoire de travail sur “l’emplacement du fichier source” (l’emplacement où votre session R actuelle est sauvegardée). S’ils se trouvent dans des dossiers différents, cliquez sur l’option “choisir un répertoire” et recherchez l’emplacement du jeu de données téléchargé.
Vous pouvez également effectuer cette opération en exécutant le code suivant
Une fois que vous avez défini votre répertoire de travail, manuellement ou par code, la console ci-dessous affiche le répertoire complet de votre dossier.
Lisez l’ensemble de données téléchargé sous le nom de “cueingData” et effectuez les exercices qui l’accompagnent au mieux de vos capacités.
# Read xlsx file
cueingData = read_excel("ProactiveControlCueing.xlsx")
Fichiers à télécharger :
- ProactiveControlCueing.xlsx
An interactive H5P element has been excluded from this version of the text. You can view it online here:
https://ecampusontario.pressbooks.pub/rspnc/?p=144#h5p-1
Solutions
Exercice 1 – Préparation et exploration des données
Après avoir configuré les progiciels nécessaires, établi votre répertoire de travail et chargé l’ensemble de données (“cueingData”) dans RStudio, procédez aux exercices ci-dessous. Copiez et collez votre code R dans les zones de texte prévues à cet effet. Vous pouvez exporter les exercices et vos réponses à la fin de cet exercice en tant que fichier docx à partir de la page d’exportation de documents une fois que vous avez terminé.
Une fois les exercices terminés, comparez vos solutions au corrigé inclus ci-dessous. N’oubliez pas que RStudio peut produire des résultats identiques par le biais de différentes méthodes. Ne vous découragez donc pas si votre code diffère du corrigé, à condition que vos résultats soient corrects.
- Affichez les premières rangées de vos données
head(cueingData) #Displaying the first few rows
## # A tibble: 6 × 6
## ID CUE_TYPE TRIAL_TYPE SOA T1Score T2Score
## <dbl> <chr> <chr> <dbl> <dbl> <dbl>
## 1 1 INFORMATIVE NS 233 100 94.6
## 2 2 INFORMATIVE NS 233 100 97.2
## 3 3 INFORMATIVE NS 233 89.2 93.9
## 4 4 INFORMATIVE NS 233 100 91.9
## 5 5 INFORMATIVE NS 233 100 100
## 6 6 INFORMATIVE NS 233 100 97.3
2. Définissez vos facteurs et vérifiez leur structure. Assurez-vous que vos mesures dépendantes sont sous forme numérique et que vos facteurs et niveaux sont correctement configurés.
cueingData <- cueingData %>%
convert_as_factor(ID, CUE_TYPE, TRIAL_TYPE, SOA) #setting up factors
str(cueingData) #checking that factors and levels are set-up correctly. Checking to see that dependent measures are in numerical format.
## # A tibble: 6 × 6
## ID CUE_TYPE TRIAL_TYPE SOA T1Score T2Score
## <dbl> <chr> <chr> <dbl> <dbl> <dbl>
## 1 1 INFORMATIVE NS 233 100 94.6
## 2 2 INFORMATIVE NS 233 100 97.2
## 3 3 INFORMATIVE NS 233 89.2 93.9
## 4 4 INFORMATIVE NS 233 100 91.9
## 5 5 INFORMATIVE NS 233 100 100
## 6 6 INFORMATIVE NS 233 100 97.3
## tibble [192 × 6] (S3: tbl_df/tbl/data.frame)
## $ ID : Factor w/ 16 levels "1","2","3","4",..: 1 2 3 4 5 6 7 8 9 10 ...
## $ CUE_TYPE : Factor w/ 2 levels "INFORMATIVE",..: 1 1 1 1 1 1 1 1 1 1 ...
## $ TRIAL_TYPE: Factor w/ 2 levels "NS","S": 1 1 1 1 1 1 1 1 1 1 ...
## $ SOA : Factor w/ 3 levels "233","467","700": 1 1 1 1 1 1 1 1 1 1 ...
## $ T1Score : num [1:192] 100 100 89.2 100 100 ...
## $ T2Score : num [1:192] 94.6 97.2 93.9 91.9 100 ...
3. Effectuer des contrôles de base des données pour vérifier les valeurs manquantes et la cohérence des données
sum(is.na(cueingData)) # Checking for missing values in the dataset
summary(cueingData) # Viewing the summary of the dataset to check for inconsistencies
## ID CUE_TYPE TRIAL_TYPE SOA T1Score
## 1 : 12 INFORMATIVE :96 NS:96 233:64 Min. : 32.43
## 2 : 12 UNINFORMATIVE:96 S :96 467:64 1st Qu.: 77.78
## 3 : 12 700:64 Median : 95.87
## 4 : 12 Mean : 86.33
## 5 : 12 3rd Qu.:100.00
## 6 : 12 Max. :100.00
## (Other):120
## T2Score
## Min. : 29.63
## 1st Qu.: 83.97
## Median : 95.76
## Mean : 87.84
## 3rd Qu.:100.00
## Max. :100.00
- Vos données correspondent-elles à un plan équilibré ou déséquilibré ? (Conseil : utilisez un code pour indiquer le nombre d’observations par combinaison de facteurs)
table(cueingData$CUE_TYPE, cueingData$TRIAL_TYPE, cueingData$SOA) #checking the number of observations per condition or combination of factors. Data is a balanced design since there is an equal number of observations per cell.
## , , = 233
##
##
## NS S
## INFORMATIVE 16 16
## UNINFORMATIVE 16 16
##
## , , = 467
##
##
## NS S
## INFORMATIVE 16 16
## UNINFORMATIVE 16 16
##
## , , = 700
##
##
## NS S
## INFORMATIVE 16 16
## UNINFORMATIVE 16 16
Exercice 2 – Calculer les statistiques sommaires
Nous utiliserons ici la bibliothèque Superb pour calculer nos statistiques sommaires avec l’erreur standard des mesures moyennes qui ont été corrigées pour les comparaisons entre sujets.
Pour vous familiariser avec la bibliothèque Superb, je vous suggère de lire l’article publié suivant et de regarder les tutoriels YouTube.
Cousineau D, Goulet M, Harding B (2021). “Graphiques sommaires avec barres d’erreur ajustées : The superb framework with an implementation in R.” Advances in Methods and Practices in Psychological Science, 2021, 1-46. doi : https://doi.org/10.1177/25152459211035109
Walker, J. A. L. (2021). “Summary plots with adjusted error bars (superb)”. Vidéo Youtube, accessible ici.
Walker, J. A. L. (2021). Summary plots with adjusted error bars (superb). Extrait de https://www.youtube.com/watch?v=rw_6ll5nVus
Pour jouer avec les différentes fonctionnalités de la bibliothèque superb, une application Shiny avec une vue de constructeur pour la bibliothèque est également disponible sur le web. Vous trouverez également ci-dessous une ressource utile pour naviguer dans le code R de la bibliothèque Superb.
5. La bibliothèque Superb exige que votre jeu de données soit dans un format large. Convertissez donc votre jeu de données d’un format long à un format large. Enregistrez-le sous “cueingData.wide”.
cueingData.wide <- cueingData %>%
pivot_wider(names_from = c(TRIAL_TYPE, SOA, CUE_TYPE),
values_from = c(T1Score, T2Score) )
6. En utilisant superbPlot() et cueingData.wide, calculez la moyenne et l’erreur standard de la moyenne (SEM) pour les scores T1 et T2 à chaque niveau des facteurs. Veillez à calculer les valeurs SEM corrigées de Cousineau-Morey.
- Vous devez le faire séparément pour chacune de vos mesures dépendantes. Enregistrez votre fonction superbplot pour T1Score sous “EXP1.T1.plot” et sous “EXP1.T2.plot” pour T2Score.
- Renommez les niveaux des facteurs dans chaque graphique. Actuellement, les niveaux sont numérotés. Nous voulons que les niveaux de SOA soient 233, 467 et 700 ; que les niveaux de type de repère soient Informatif et Non informatif, et que les niveaux de type d’essai soient Sélection et Pas de sélection (Astuce : pour accéder aux données récapitulatives, utilisez EXP1.T1.plot$data$insertfactorname).
EXP1.T1.plot <- superbPlot(cueingData.wide,
WSFactors = c("SOA(3)", "CueType(2)", "TrialType(2)"),
variables = c("T1Score_NS_233_INFORMATIVE", "T1Score_NS_467_INFORMATIVE",
"T1Score_NS_700_INFORMATIVE", "T1Score_NS_233_UNINFORMATIVE",
"T1Score_NS_467_UNINFORMATIVE", "T1Score_NS_700_UNINFORMATIVE",
"T1Score_S_233_INFORMATIVE", "T1Score_S_467_INFORMATIVE",
"T1Score_S_700_INFORMATIVE", "T1Score_S_233_UNINFORMATIVE",
"T1Score_S_467_UNINFORMATIVE", "T1Score_S_700_UNINFORMATIVE"),
statistic = "mean",
errorbar = "SE",
adjustments = list(
purpose = "difference",
decorrelation = "CM",
popSize = 32
),
plotStyle = "line",
factorOrder = c("SOA", "CueType", "TrialType"),
lineParams = list(size=1, linetype="dashed"),
pointParams = list(size = 3))
## superb::FYI: Here is how the within-subject variables are understood:
## SOA CueType TrialType variable
## 1 1 1 T1Score_NS_233_INFORMATIVE
## 2 1 1 T1Score_NS_467_INFORMATIVE
## 3 1 1 T1Score_NS_700_INFORMATIVE
## 1 2 1 T1Score_NS_233_UNINFORMATIVE
## 2 2 1 T1Score_NS_467_UNINFORMATIVE
## 3 2 1 T1Score_NS_700_UNINFORMATIVE
## 1 1 2 T1Score_S_233_INFORMATIVE
## 2 1 2 T1Score_S_467_INFORMATIVE
## 3 1 2 T1Score_S_700_INFORMATIVE
## 1 2 2 T1Score_S_233_UNINFORMATIVE
## 2 2 2 T1Score_S_467_UNINFORMATIVE
## 3 2 2 T1Score_S_700_UNINFORMATIVE
## superb::FYI: The HyunhFeldtEpsilon measure of sphericity per group are 0.134
## superb::FYI: Some of the groups' data are not spherical. Use error bars with caution.
EXP1.T2.plot <- superbPlot(cueingData.wide,
WSFactors = c("SOA(3)", "CueType(2)", "TrialType(2)"),
variables = c("T2Score_NS_233_INFORMATIVE", "T2Score_NS_467_INFORMATIVE",
"T2Score_NS_700_INFORMATIVE", "T2Score_NS_233_UNINFORMATIVE",
"T2Score_NS_467_UNINFORMATIVE", "T2Score_NS_700_UNINFORMATIVE",
"T2Score_S_233_INFORMATIVE", "T2Score_S_467_INFORMATIVE",
"T2Score_S_700_INFORMATIVE", "T2Score_S_233_UNINFORMATIVE",
"T2Score_S_467_UNINFORMATIVE", "T2Score_S_700_UNINFORMATIVE"),
statistic = "mean",
errorbar = "SE",
adjustments = list(
purpose = "difference",
decorrelation = "CM",
popSize = 32
),
plotStyle = "line",
factorOrder = c("SOA", "CueType", "TrialType"),
lineParams = list(size=1, linetype="dashed"),
pointParams = list(size = 3)
)
## superb::FYI: Here is how the within-subject variables are understood:
## SOA CueType TrialType variable
## 1 1 1 T2Score_NS_233_INFORMATIVE
## 2 1 1 T2Score_NS_467_INFORMATIVE
## 3 1 1 T2Score_NS_700_INFORMATIVE
## 1 2 1 T2Score_NS_233_UNINFORMATIVE
## 2 2 1 T2Score_NS_467_UNINFORMATIVE
## 3 2 1 T2Score_NS_700_UNINFORMATIVE
## 1 1 2 T2Score_S_233_INFORMATIVE
## 2 1 2 T2Score_S_467_INFORMATIVE
## 3 1 2 T2Score_S_700_INFORMATIVE
## 1 2 2 T2Score_S_233_UNINFORMATIVE
## 2 2 2 T2Score_S_467_UNINFORMATIVE
## 3 2 2 T2Score_S_700_UNINFORMATIVE
## superb::FYI: The HyunhFeldtEpsilon measure of sphericity per group are 0.226
## superb::FYI: Some of the groups' data are not spherical. Use error bars with caution.
# Re-naming levels of the factors
levels(EXP1.T1.plot$data$SOA) <- c("1" = "233", "2" = "467", "3" = "700")
levels(EXP1.T2.plot$data$SOA) <- c("1" = "233", "2" = "467", "3" = "700")
levels(EXP1.T1.plot$data$TrialType) <- c("1" = "No Selection", "2" = "Selection")
levels(EXP1.T2.plot$data$TrialType) <- c("1" = "No Selection", "2" = "Selection")
levels(EXP1.T1.plot$data$CueType) <- c("1" = "Informative", "2" = "Uninformative")
levels(EXP1.T2.plot$data$CueType) <- c("1" = "Informative", "2" = "Uninformative")
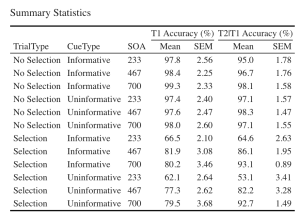
7. Créons un magnifique tableau HTML imprimable des statistiques récapitulatives pour les scores T1 et T2. Ce tableau récapitulatif peut ensuite être utilisé dans votre manuscrit. Je vous suggère de visiter le lien suivant pour obtenir des guides sur la façon de créer des tableaux imprimables. Personnalisation du tableau HTML
- Commencez par extraire les données de statistiques sommaires avec les moyennes de groupe et les valeurs SEM de CousineauMorey de chaque fonction de tracé et enregistrez-les en tant que cadre de données séparément pour les données T1 et T2 (vous devriez avoir deux cadres de données avec vos statistiques sommaires nommés “EXP1.T1.summaryData” et “EXP1.T2.summaryData”).
- Dans vos deux cadres de données avec les statistiques sommaires, arrondissez vos moyennes à une décimale et vos valeurs SEM à deux décimales.
- Fusionnez les données récapitulatives de T1Score et T2Score et enregistrez-les sous “EXP1_summarystat_results”
- Dans ce tableau fusionné, supprimez les colonnes contenant les valeurs SEM négatives (valeurs SEM de largeur inférieure).
- Renommez les colonnes de ce cadre de données fusionné de sorte que le nom des colonnes contenant les moyennes T1Score et T2Score soit “Means” et que les colonnes contenant les scores SEM pour l’une ou l’autre des variables dépendantes soient “SEM”.
- Intitulez votre tableau “Statistiques sommaires”
- Réglez la police de votre texte sur “Cambria” et la taille de la police sur 14.
- Définissez les en-têtes des colonnes T1Score means et SEM comme “T1 Accuracy (%)”.
- Définissez les en-têtes des colonnes T2Score means et SEM comme “T2 Accuracy (%)”.
# Extracting summary data with CousineauMorey SEM Bars
EXP1.T1.summaryData <- data.frame(EXP1.T1.plot$data)
EXP1.T2.summaryData <- data.frame(EXP1.T2.plot$data)
# Rounding values in each column
# round(x, 1) rounds to the specified number of decimal places
EXP1.T1.summaryData$center <- round(EXP1.T1.summaryData$center,1)
EXP1.T1.summaryData$upperwidth <- round(EXP1.T1.summaryData$upperwidth,2)
EXP1.T2.summaryData$center <- round(EXP1.T2.summaryData$center,1)
EXP1.T2.summaryData$upperwidth <- round(EXP1.T2.summaryData$upperwidth,2)
# merging T1 and T2|T1 summary tables
EXP1_summarystat_results <- merge(EXP1.T1.summaryData, EXP1.T2.summaryData, by=c("TrialType","CueType","SOA"))
# Rename the column name
colnames(EXP1_summarystat_results)[colnames(EXP1_summarystat_results) == "center.x"] ="Mean"
colnames(EXP1_summarystat_results)[colnames(EXP1_summarystat_results) == "center.y"] ="Mean"
colnames(EXP1_summarystat_results)[colnames(EXP1_summarystat_results) == "upperwidth.x"] ="SEM"
colnames(EXP1_summarystat_results)[colnames(EXP1_summarystat_results) == "upperwidth.y"] ="SEM"
# deleting columns by name "lowerwidth.x" and "lowerwidth.y" in each summary table
EXP1_summarystat_results <- EXP1_summarystat_results[ , ! names(EXP1_summarystat_results) %in% c("lowerwidth.x", "lowerwidth.y")]
#removing suffixes from column names
colnames(EXP1_summarystat_results)<-gsub(".1","",colnames(EXP1_summarystat_results))
# Printable ANOVA html
EXP1_summarystat_results %>%
kbl(caption = "Summary Statistics") %>%
kable_classic(full_width = F, html_font = "Cambria", font_size = 14) %>%
add_header_above(c(" " = 3, "T1 Accuracy (%)" = 2, "T2|T1 Accuracy (%)" = 2))
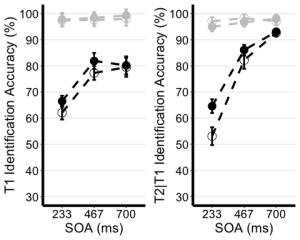
Exercice 3 – Visualiser les données
8. Utilisez le tableau de statistiques récapitulatives non édité de l’exercice 2 (EXP1.T1.summaryData et EXP1.T3.summaryData) et la fonction ggplot() pour créer des graphiques linéaires récapitulatifs distincts pour les scores T1 et T2. Le graphique linéaire visualise la relation entre la SOA et la mesure dépendante tout en tenant compte des facteurs de type de repère et de type d’essai. Votre graphique doit présenter les caractéristiques suivantes :
- Tracer le SOA sur l’axe des abscisses et l’intituler “SOA (ms)”
- Tracez la mesure dépendante sur l’axe des ordonnées et intitulez l’axe des ordonnées “Précision d’identification T1” pour le tracé T1, et “Précision d’identification T2|T1 (%)” pour le tracé T2.
- Définissez la couleur de vos lignes en fonction du type d’essai.
- Définissez la forme des points pour chaque valeur de la ligne par type d’indice.
- Utilisez la fonction geom_point() pour personnaliser la forme de vos points. Utilisez des cercles pleins pour les indices informatifs et des cercles creux pour les indices non informatifs.
- Utilisez la fonction scale_color_manual() pour personnaliser les couleurs des lignes. Réglez la couleur des lignes représentant les essais de sélection sur “black” et celle des lignes représentant les essais de non-sélection sur “gray78”.
- Utilisez geom_line() pour personnaliser le type de ligne. Réglez le type de ligne sur pointillé et la largeur de ligne sur 1,2.
- Personnalisez votre axe des y. Fixez la valeur minimale à 30 et la valeur maximale à 100.
- Utilisez la fonction scale_y_continuous() pour que les valeurs de l’axe des y augmentent par incréments de 10.
- Utilisez la fonction geom_errorbar() pour tracer des barres d’erreur en utilisant les valeurs SEM calculées dans le tableau récapitulatif.
- Définissez le thème du tracé sur theme_classic()
- Utilisez la fonction theme() pour personnaliser la taille de la police de l’axe des x et la largeur des lignes. Modifier la taille de la police du titre principal de l’axe à 16, et les étiquettes de l’axe des x à 14.
- Ajoutez des lignes de grille horizontales.
- Ne pas inclure de légende.
- Stockez vos deux graphiques sous les noms “T1.ggplotplot” et “T2.ggplotplot”
EXP1.T1.ggplotplot <- ggplot(EXP1.T1.summaryData, aes(x=SOA, y=center, color=TrialType, shape = CueType,
group=interaction(CueType, TrialType))) +
geom_point(data=filter(EXP1.T1.summaryData, CueType == "Uninformative"), shape=1, size=4.5) + # assigning shape type to level of factor
geom_point(data=filter(EXP1.T1.summaryData, CueType == "Informative"), shape=16, size=4.5) + # assigning shape type to level of factor
geom_line(linetype="dashed", linewidth=1.2) + # change line thickness and line style
scale_color_manual(values = c("gray78", "black") ) +
xlab("SOA (ms)") +
ylab("T1 Identification Accuracy (%)") +
theme_classic() + # It has no background, no bounding box.
theme(axis.line=element_line(size=1.5), # We make the axes thicker...
axis.text = element_text(size = 14, colour = "black"), # their text bigger...
axis.title = element_text(size = 16, colour = "black"), # their labels bigger...
panel.grid.major.y = element_line(), # adding horizontal grid lines
legend.position = "none") +
coord_cartesian(ylim=c(30, 100)) +
scale_y_continuous(breaks=seq(30, 100, 10)) + # Ticks from 30-100, every 10
geom_errorbar(aes(ymin=center+lowerwidth, ymax=center+upperwidth), width = 0.12, size = 1) # adding error bars from summary table
EXP1.T2.ggplotplot <- ggplot(EXP1.T2.summaryData, aes(x=SOA, y=center, color=TrialType, shape=CueType,
group=interaction(CueType, TrialType))) +
geom_point(data=filter(EXP1.T2.summaryData, CueType == "Uninformative"), shape=1, size=4.5) + # assigning shape type to level of factor
geom_point(data=filter(EXP1.T2.summaryData, CueType == "Informative"), shape=16, size=4.5) + # assigning shape type to level of factor
geom_line(linetype="dashed", linewidth=1.2) + # change line thickness and line style
scale_color_manual(values = c("gray78", "black")) +
xlab("SOA (ms)") +
ylab("T2|T1 Identification Accuracy (%)") +
theme_classic() + # It has no background, no bounding box.
theme(axis.line=element_line(size=1.5), # We make the axes thicker...
axis.text = element_text(size = 14, colour = "black"), # their text bigger...
axis.title = element_text(size = 16, colour = "black"), # their labels bigger...
panel.grid.major.y = element_line(), # adding horizontal grid lines
legend.position = "none") +
guides(fill = guide_legend(override.aes = list(shape = 16) ),
shape = guide_legend(override.aes = list(fill = "black"))) +
coord_cartesian(ylim=c(30, 100)) +
scale_y_continuous(breaks=seq(30, 100, 10)) + # Ticks from 30-100, every 10
geom_errorbar(aes(ymin=center+lowerwidth, ymax=center+upperwidth), width = 0.12, size = 1) # adding error bars from summary table
9. Utilisez ggarrange() pour afficher vos tracés ensemble.
ggarrange(EXP1.T1.ggplotplot, EXP1.T2.ggplotplot,
nrow = 1, ncol = 2, common.legend = F,
widths = 8, heights = 5)
Exercice 4 – L’analyse principale
Étant donné que les performances des essais de non-sélection sont proches du plafond (près de 100 %), nous concentrerons nos analyses sur les essais de sélection.
10. Utilisez les données en format long (“cueingData”) et la fonction anova_test(), et calculez une ANOVA à deux voies pour chaque variable dépendante, mais sur les essais de sélection uniquement. Définissez le type de repère et l’AOS comme facteurs internes aux participants. (Conseil : utilisez la fonction filter())
- Réglez votre mesure de l’ampleur de l’effet sur l’éta quadratique partiel (pes).
- Assurez-vous de générer le tableau ANOVA détaillé.
- Enregistrez vos calculs et “T1_2anova” et “T2_2anova”.
- Utilisez la fonction get_anova_table() pour afficher vos tableaux d’ANOVA.
T1_2anova <- anova_test(
data = filter(cueingData, TRIAL_TYPE == "S"), dv = T1Score, wid = ID,
within = c(CUE_TYPE, SOA), detailed = TRUE, effect.size = "pes")
T2_2anova <- anova_test(
data = filter(cueingData, TRIAL_TYPE == "S"), dv = T2Score, wid = ID,
within = c(CUE_TYPE, SOA), detailed = TRUE, effect.size = "pes")
get_anova_table(T1_2anova)
## ANOVA Table (type III tests)
##
## Effect DFn DFd SSn SSd F p p<.05 pes
## 1 (Intercept) 1.00 15.00 534043.211 30705.523 260.886 6.80e-11 * 0.946
## 2 CUE_TYPE 1.00 15.00 253.665 506.454 7.513 1.50e-02 * 0.334
## 3 SOA 1.29 19.35 5091.853 2660.275 28.710 1.16e-05 * 0.657
## 4 CUE_TYPE:SOA 2.00 30.00 77.140 1061.051 1.091 3.49e-01 0.068
get_anova_table(T2_2anova)
## ANOVA Table (type III tests)
##
## Effect DFn DFd SSn SSd F p p<.05 pes
## 1 (Intercept) 1 15 593913.106 11288.706 789.169 2.19e-14 * 0.981
## 2 CUE_TYPE 1 15 661.761 426.947 23.250 2.24e-04 * 0.608
## 3 SOA 2 30 20001.563 3852.629 77.875 1.33e-12 * 0.838
## 4 CUE_TYPE:SOA 2 30 519.154 1298.144 5.999 6.00e-03 * 0.286
Exercice 5 – Les tests post-hoc
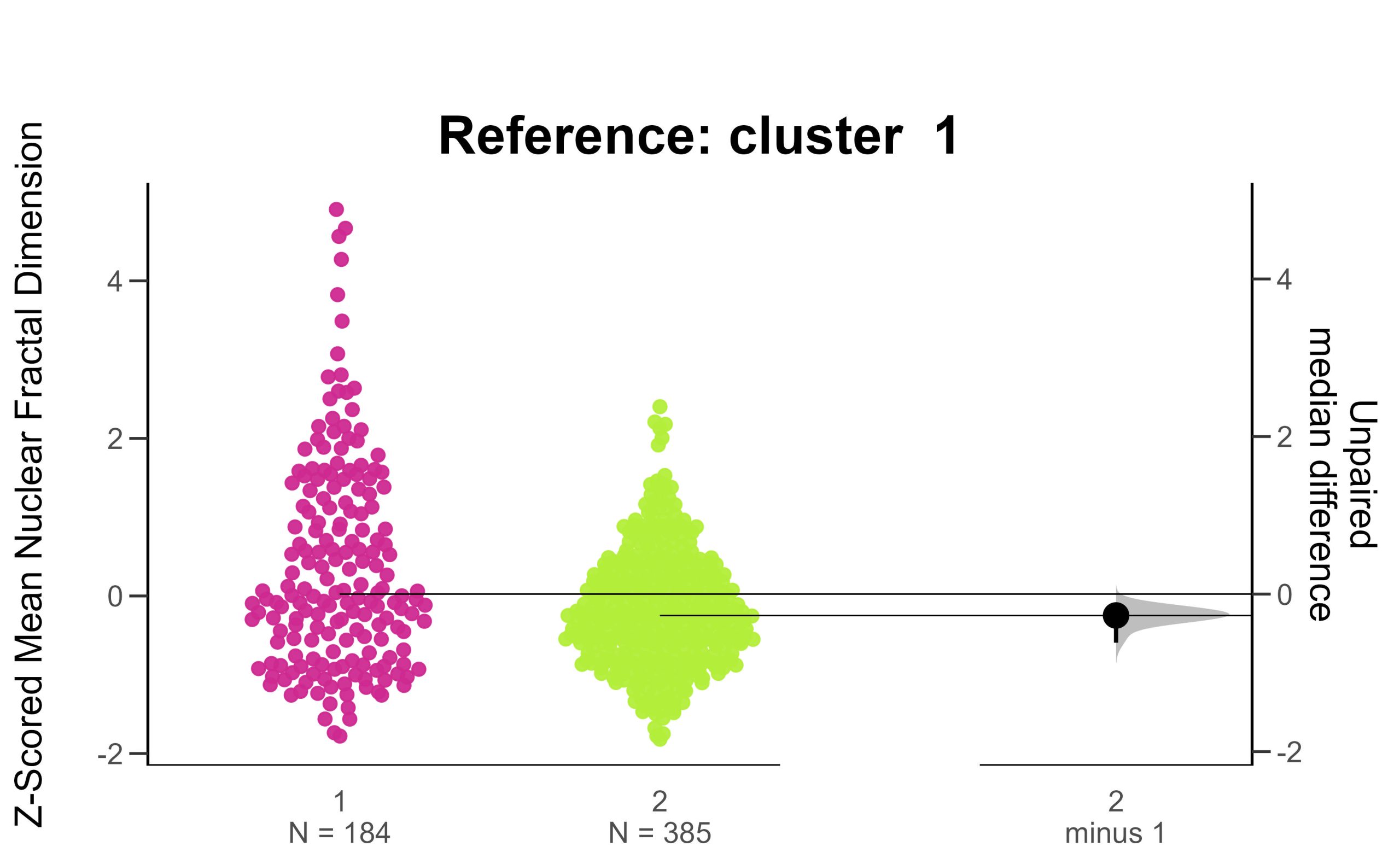
S’il existe une interaction bidirectionnelle significative, cela signifie que l’impact d’un facteur est influencé par un autre facteur. Cela signifie que les deux variables indépendantes interagissent l’une avec l’autre pour produire un effet significatif sur la variable dépendante. Étant donné qu’il existe une interaction bidirectionnelle significative entre le type de repère et l’AOS pour les scores T2, nous devons procéder à des tests post hoc afin d’explorer et de comprendre les différences spécifiques entre les niveaux ou les conditions des facteurs impliqués dans l’interaction. Cela peut nous aider à identifier les combinaisons spécifiques à l’origine de l’effet d’interaction.
11. Filtrez d’abord les essais de sélection, puis regroupez vos données par SOA et utilisez la fonction pairwise_t_test() pour comparer les essais informatifs et non informatifs à chaque niveau de SOA, stockez et affichez votre calcul sous la forme “T2_sel_pwc”.
T2_sel_pwc <- filter(cueingData, TRIAL_TYPE == "S") %>%
group_by(SOA) %>%
pairwise_t_test(T2Score ~ CUE_TYPE, paired = TRUE, p.adjust.method = "holm", detailed = TRUE) %>%
add_significance("p.adj")
T2_sel_pwc <- get_anova_table(T2_sel_pwc)
T2_sel_pwc
## # A tibble: 3 × 16
## SOA estimate .y. group1 group2 n1 n2 statistic p df
## <fct> <dbl> <chr> <chr> <chr> <int> <int> <dbl> <dbl> <dbl>
## 1 233 11.5 T2Score INFORMATIVE UNINFO… 16 16 4.36 5.55e-4 15
## 2 467 3.89 T2Score INFORMATIVE UNINFO… 16 16 1.97 6.8 e-2 15
## 3 700 0.356 T2Score INFORMATIVE UNINFO… 16 16 0.190 8.52e-1 15
## # ℹ 6 more variables: conf.low <dbl>, conf.high <dbl>, method <chr>,
## # alternative <chr>, p.adj <dbl>, p.adj.signif <chr>
Remarques finales
Superbe travail !
Vous avez réussi à reconstruire une figure et à effectuer des analyses dans le cadre d’un projet de mémoire de maîtrise. Les données sur lesquelles vous avez travaillé ont été recueillies dans le laboratoire d’attention et de mémoire du département de psychologie, de neuroscience et de comportement (PNB) de l’Université McMaster. Ce laboratoire prospère sous l’égide du Dr Bruce Milliken, dont les recherches sont profondément ancrées dans le domaine de la cognition humaine. L’objectif principal du laboratoire est d’élucider les processus fondamentaux qui sous-tendent l’attention, la mémoire et le contrôle cognitif. L’étendue des recherches menées ici englobe un large éventail de sujets. Il s’agit notamment d’étudier les nuances qui différencient les processus mentaux conscients et inconscients, d’explorer la manière dont les mécanismes attentionnels influencent et soutiennent l’apprentissage et la mémoire, de comprendre le rôle de l’imagerie visuelle dans la formation de l’attention et de la perception, et d’examiner l’impact de l’apprentissage implicite sur l’orchestration du contrôle de l’attention. Chaque projet au sein de ce laboratoire témoigne de l’engagement à faire progresser notre compréhension de la tapisserie complexe de la cognition humaine.
Références et lectures complémentaires :
Si vous souhaitez approfondir l’ensemble des données et les spécificités du projet de thèse, je vous encourage à explorer la thèse de doctorat librement accessible ou l’article publié cité ci-dessous :
Montakhaby Nodeh, S., MacLellan, E., & Milliken, B. (2024). Proactive control: Endogenous cueing effects in a two-target attentional blink task. Consciousness and Cognition, 118, 103648. Elsevier BV. https://doi.org/10.1016/j.concog.2024.103648
P02: Laboratoire de cognition et de mémoire
Sevda Montakhaby Nodeh
Laboratoire de cognition et de mémoire
Bienvenue ! Dans le cadre de cette tâche, nous allons entrer dans un laboratoire de cognition et de mémoire de l’Université McMaster. Plus précisément, nous examinerons les données d’une étude fascinante de psychologie cognitive qui explore le rôle de la répétition dans la mémoire de reconnaissance.
La plupart d’entre nous connaissent l’expression “c’est en forgeant qu’on devient forgeron”. Cette expression motivante correspond à l’intuition et est confirmée par de nombreuses observations dans le monde réel. De nombreuses recherches empiriques soutiennent également ce point de vue : les occasions répétées d’encoder un stimulus améliorent la récupération ultérieure de la mémoire et l’identification perceptuelle. Ces observations suggèrent que la répétition d’un stimulus renforce les représentations sous-jacentes dans la mémoire.
La présente étude se concentre sur une idée contradictoire, à savoir que la répétition d’un stimulus peut affaiblir l’encodage de la mémoire. L’expérience comprenait trois étapes : une phase d’étude, une phase de distraction et un test de mémoire de reconnaissance surprise.
La présente étude se concentre sur une idée contradictoire, à savoir que la répétition du stimulus peut affaiblir l’encodage de la mémoire. L’expérience comprenait trois étapes : une phase d’étude, une phase de distraction et un test de mémoire de reconnaissance par surprise.
Dans la phase d’étude, les participants prononcent à haute voix un mot cible rouge précédé d’un mot premier vert brièvement présenté. Sur la moitié des essais, le mot principal et le mot cible étaient identiques (essais répétés), et sur l’autre moitié des essais, le mot principal et le mot cible étaient différents (essais non répétés). La figure ci-dessous donne un aperçu des deux types d’essais. Après la phase d’étude, les participants se sont livrés à une tâche de distraction de 10 minutes consistant en des problèmes mathématiques qu’ils devaient résoudre à la main.
La phase finale consistait en un test de mémoire de reconnaissance surprise où, à chaque essai, on leur montrait un mot rouge et on leur demandait de répondre “ancien” si le mot du test était un mot qu’ils avaient déjà vu à l’étude, et “nouveau” s’ils n’avaient jamais rencontré ce mot auparavant. La moitié des essais du test étaient des mots de la phase d’étude et l’autre moitié des mots nouveaux.
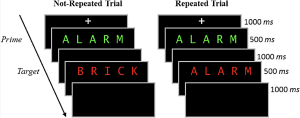
Commençons par exécuter le code suivant pour charger les bibliothèques requises. Veillez à lire les commentaires intégrés dans le code pour comprendre ce que fait chaque ligne de code.
Remarque : les cases grisées contiennent le code R, le signe “#” indiquant un commentaire qui ne s’exécutera pas dans RStudio
# Load necessary libraries
library(rstatix) #for performing basic statistical tests
library(dplyr) #for sorting data
library(readxl) #for reading excel files
library(tidyr) #for data sorting and structure
library(ggplot2) #for visualizing your data
library(plotrix) #for computing basic summary stats
Assurez-vous d’avoir téléchargé l’ensemble de données requis (“RepDecrementdataset.xlsx”) pour cet exercice. Définissez le répertoire de travail de votre session R actuelle dans le dossier contenant l’ensemble de données téléchargé. Vous pouvez le faire manuellement dans le studio R en cliquant sur l’onglet “Session” en haut de l’écran, puis en cliquant sur “Set Working Directory”.
Si le fichier de données téléchargé et votre session R se trouvent dans le même dossier, vous pouvez choisir de définir votre répertoire de travail sur “l’emplacement du fichier source” (l’emplacement où votre session R actuelle est sauvegardée). S’ils se trouvent dans des dossiers différents, cliquez sur l’option “choisir un répertoire” et recherchez l’emplacement du jeu de données téléchargé.
Vous pouvez également effectuer cette opération en exécutant le code suivant
Une fois que vous avez défini votre répertoire de travail, manuellement ou par code, la console ci-dessous affiche le répertoire complet de votre dossier.
Lisez l’ensemble de données téléchargé en tant que “MemoryData” et effectuez les exercices qui l’accompagnent au mieux de vos capacités.
MemoryData <- read_excel('RepDecrementdataset.xlsx')
Fichiers à télécharger :
- RepDecrementdataset.xlsx
An interactive H5P element has been excluded from this version of the text. You can view it online here:
https://ecampusontario.pressbooks.pub/rspnc/?p=148#h5p-2
Solutions
Exercice 1 – Préparation et exploration des données
Remarque : les cases grisées contiennent le code R, tandis que les cases blanches affichent la sortie du code, telle qu’elle apparaît dans RStudio.
Le signe “#” indique un commentaire qui ne sera pas exécuté dans RStudio.
1. Affichez les premières lignes de votre jeu de données pour vous familiariser avec sa structure et son contenu.
head(MemoryData) #Displaying the first few rows
## # A tibble: 6 × 7
## ID Hits_NRep Hits_Rep FalseAlarms Misses_Nrep Misses_Rep CorrectRej
## <dbl> <dbl> <dbl> <dbl> <dbl> <dbl> <dbl>
## 1 1 46 34 13 14 26 107
## 2 2 43 44 27 17 16 93
## 3 3 43 35 23 17 24 97
## 4 4 37 36 56 23 24 64
## 5 5 39 35 49 21 25 71
## 6 6 38 43 28 22 17 92
str(MemoryData) #Checking structure of dataset
## tibble [24 × 7] (S3: tbl_df/tbl/data.frame)
## $ ID : num [1:24] 1 2 3 4 5 6 7 8 9 10 ...
## $ Hits_NRep : num [1:24] 46 43 43 37 39 38 20 24 36 38 ...
## $ Hits_Rep : num [1:24] 34 44 35 36 35 43 11 29 43 27 ...
## $ FalseAlarms: num [1:24] 13 27 23 56 49 28 4 11 46 9 ...
## $ Misses_Nrep: num [1:24] 14 17 17 23 21 22 40 36 23 22 ...
## $ Misses_Rep : num [1:24] 26 16 24 24 25 17 49 31 17 33 ...
## $ CorrectRej : num [1:24] 107 93 97 64 71 92 116 109 74 111 ...
## [1] "ID" "Hits_NRep" "Hits_Rep" "FalseAlarms" "Misses_Nrep"
## [6] "Misses_Rep" "CorrectRej"
2. Calculer le nombre total d’essais pour chaque condition :
- (a) Pour chaque participant, additionnez le nombre de réponses positives pour les essais non répétés et les essais non répétés manqués. Enregistrez ce total dans une nouvelle colonne intitulée “TotalNRep”. La valeur doit être de 60 pour tous les participants, ce qui correspond au nombre total de types d’essais non répétés.
- (b) Répétez le processus pour les essais répétés, en enregistrant la somme dans “TotalRep” (60 essais).
- (c) De même, additionnez le nombre de fausses alarmes et de rejets corrects pour représenter le nombre total de nouveaux essais (120 essais) et enregistrez cette somme dans “TotalNew”.
Notez que si la valeur de “TotalNRep” et “TotalRep” est inférieure à 60 pour un participant, cela indique que certains essais de mots ont été exclus pendant la phase d’étude en raison de problèmes (par exemple, le participant a lu à haute voix le mot principal au lieu du mot cible, ce qui a entraîné la détérioration de l’essai).
MemoryData <- MemoryData %>%
mutate(TotalNRep = Hits_NRep + Misses_Nrep)
MemoryData <- MemoryData %>%
mutate(TotalRep = Hits_Rep + Misses_Rep)
MemoryData <- MemoryData %>%
mutate(TotalNew = FalseAlarms + CorrectRej)
3. Transformez les nombres des colonnes “hits”, “misses”, “false alarms” et “correct rejections” en proportions. Pour ce faire, divisez chaque nombre par le nombre total d’essais pour la condition concernée (par exemple, divisez les réponses positives pour les essais non répétés par “TotalNRep”).
MemoryData$Hits_NRep <- (MemoryData$Hits_NRep/MemoryData$TotalNRep)
MemoryData$Misses_Nrep <- (MemoryData$Misses_Nrep/MemoryData$TotalNRep)
MemoryData$Hits_Rep <- (MemoryData$Hits_Rep/MemoryData$TotalRep)
MemoryData$Misses_Rep <- (MemoryData$Misses_Rep/MemoryData$TotalRep)
MemoryData$CorrectRej <- (MemoryData$CorrectRej/MemoryData$TotalNew)
MemoryData$FalseAlarms <- (MemoryData$FalseAlarms/MemoryData$TotalNew)
4. Une fois les proportions calculées, supprimez les colonnes “TotalNew”, “TotalRep” et “TotalNRep” de l’ensemble de données, car elles ne sont plus nécessaires pour la suite de l’analyse.
MemoryData <- MemoryData[, !names(MemoryData) %in% c("TotalNew", "TotalRep","TotalNRep")]
5. Utilisez la fonction pivot_longer() du package tidyr pour convertir vos données du format large au format long. Effectuez un pivot des colonnes “Hits_NRep”, “Hits_Rep” et “FalseAlarms”, en définissant les nouveaux noms de colonnes “Condition” et “Proportion” pour les données remodelées.
long_df <- MemoryData %>%
pivot_longer(
cols = c(Hits_NRep, Hits_Rep, FalseAlarms),
names_to = "Condition",
values_to = "Proportion"
)
Exercice 2 : Calcul des statistiques sommaires et correction de la variabilité intra-sujet
6. À l’aide de votre ensemble de données au format long, regroupez vos données par ID et calculez la moyenne par sujet et la moyenne générale de la colonne Proportions.
- (a) Ajustez le score de chaque individu en soustrayant sa moyenne et en ajoutant la moyenne générale.
- (b) Calculez la moyenne et le SEM des scores ajustés pour chaque condition.
- (c) Utilisez les scores ajustés pour calculer le SEM intra-sujet. d.Regroupez les données par condition et calculez la moyenne et le SEM.
data_adjusted <- long_df %>%
group_by(ID) %>%
mutate(SubjectMean = mean(Proportion, na.rm = TRUE)) %>%
ungroup() %>%
mutate(GrandMean = mean(Proportion, na.rm = TRUE)) %>%
mutate(AdjustedScore = Proportion - SubjectMean + GrandMean)
# Calculate the mean and SEM of the adjusted scores
summary_df <- data_adjusted %>%
group_by(Condition) %>%
summarize(
AdjustedMean = mean(AdjustedScore, na.rm = TRUE),
AdjustedSEM = sd(AdjustedScore, na.rm = TRUE) / sqrt(n())
)
Exercice 3 : Visualisation des données
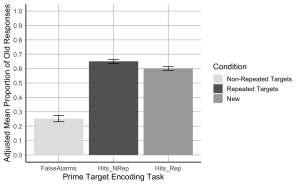
7. Créez un diagramme à barres où l’axe x représente les conditions de la tâche d’encodage de l’amorce et de la cible, l’axe y montre la proportion moyenne ajustée des anciennes réponses, et les barres d’erreur représentent le SEM ajusté. Commencez par définir des couleurs personnalisées pour chaque condition. La couleur de la barre présentant les fausses alarmes ou “New” doit être “gray89” ; la couleur de la barre “Hits_Nrep” ou “Non-Repeated Targets” doit être “gray39” ; la couleur de la barre “Hits_Rep” ou “Repeated Targets” doit être “darkgrey”.
- (a) L’axe des x doit être intitulé “tâche d’encodage de la cible principale”.
- (b) L’axe des ordonnées doit être intitulé “Proportion moyenne corrigée des anciennes réponses”
- (c) Ajoutez des barres d’erreur à chaque barre pour représenter le SEM corrigé.
- (d) Faites en sorte que les lignes des axes x et y soient noires et pleines.
- (e) Veillez à ce que le graphique soit minimaliste et ne comporte que les principales lignes de la grille.
- (f) Ajouter une légende pour indiquer les catégories de conditions. La légende doit être la suivante : cibles non répétées au lieu de “Hits_Nrep”, cibles répétées au lieu de “Hits_Rep”, et nouvelles au lieu de “Fausses alarmes”.
- (g) Fixez les valeurs minimale et maximale de l’axe des y à 0 et 1, respectivement.
- (h) Les valeurs de l’axe des y doivent augmenter de 0,1.
# Create the bar plot with adjusted SEM error bars
ggplot(summary_df, aes(x = Condition, y = AdjustedMean, fill = Condition)) +
geom_bar(stat = "identity", position = position_dodge()) +
geom_errorbar(aes(ymin = AdjustedMean - AdjustedSEM, ymax = AdjustedMean + AdjustedSEM), width = 0.2, position = position_dodge(0.9)) +
scale_fill_manual(values = c("Hits_NRep" = "gray39", "Hits_Rep" = "darkgrey", "FalseAlarms" = "gray89"),
labels = c("Non-Repeated Targets", "Repeated Targets", "New")) +
labs(
x = "Prime Target Encoding Task",
y = "Adjusted Mean Proportion of Old Responses",
fill = "Condition"
) +
scale_y_continuous(breaks = seq(0, 1, by = 0.1), limits = c(0, 1)) +
theme_minimal(base_size = 14) +
theme(
axis.line = element_line(color = "black"),
axis.title = element_text(color = "black"),
panel.grid.major = element_line(color = "grey", size = 0.5),
panel.grid.minor = element_blank(),
legend.title = element_text(color = "black")
)
Exercice 4 – L’analyse principale
8. À l’aide du fichier de données au format large “MemoryData”, effectuez un test t à deux échantillons appariés en comparant le taux de réussite cumulé dans les deux conditions de répétition (répété/non répété) au taux de fausses alarmes pour évaluer la capacité des participants à distinguer les anciens éléments des nouveaux.
- (a) Calculez le taux de réussite moyen en faisant la moyenne des taux de réussite des conditions “Hits_NRep” (non répété) et “Hits_Rep” (répété) pour chaque participant.
- (b) Effectuez un test t pour échantillons appariés afin de comparer le taux de réussite (combiné dans les deux conditions de répétition) au taux de fausses alarmes afin d’évaluer la capacité des participants à distinguer les anciens éléments des nouveaux.
collapsed_hitdata <- MemoryData %>%
mutate(HitRate = (Hits_NRep + Hits_Rep) / 2)
# Conduct paired sample t-tests
t_test_results <- t.test(collapsed_hitdata$HitRate, collapsed_hitdata$FalseAlarms, paired = TRUE)
print(t_test_results)
##
## Paired t-test
##
## data: collapsed_hitdata$HitRate and collapsed_hitdata$FalseAlarms
## t = 11.621, df = 23, p-value = 4.179e-11
## alternative hypothesis: true mean difference is not equal to 0
## 95 percent confidence interval:
## 0.3071651 0.4401983
## sample estimates:
## mean difference
## 0.3736817
#Hit rates were higher than false alarm rates, t(23) = 11.62, p < .001.
9. En utilisant le fichier de données au format large “MemoryData”, effectuez un test t à deux échantillons appariés comparant les taux de réussite pour les cibles non répétées et répétées.
# Conduct paired sample t-tests for non-repeated vs repeated hit rates
t_test_results_hits <- t.test(collapsed_hitdata$Hits_NRep, collapsed_hitdata$Hits_Rep, paired = TRUE)
# Print the results for the hit rate comparison
print(t_test_results_hits)
##
## Paired t-test
##
## data: collapsed_hitdata$Hits_NRep and collapsed_hitdata$Hits_Rep
## t = 2.5431, df = 23, p-value = 0.01817
## alternative hypothesis: true mean difference is not equal to 0
## 95 percent confidence interval:
## 0.009071364 0.088174399
## sample estimates:
## mean difference
## 0.04862288
#Hit rates were higher for not-repeated targets than for repeated targets, t(23) = 2.54, p = .018.
P03: Laboratoire de développement de la petite enfance
Sevda Montakhaby Nodeh
Laboratoire de développement de la petite enfance
Vous êtes chercheur au Centre de recherche sur le développement de la petite enfance. Votre dernier projet porte sur la manière dont les nourrissons réagissent à différentes combinaisons de visages, de races et d’émotions musicales. Plus précisément, vous vous intéressez à la question de savoir si les enfants associent des visages de leur propre race et d’autres races à des musiques de différentes valences émotionnelles (musique joyeuse et musique triste).
Votre projet a été réalisé en collaboration avec vos collègues en Chine. Alors que vous étiez responsable de la conception de votre expérience, vos collaborateurs étaient chargés de recruter les participants et de collecter les données.
Des bébés chinois (âgés de 3 à 9 mois) ont été recrutés pour participer à votre expérience. Chaque enfant a été assigné au hasard à l’une des quatre conditions visage-race + musique dans lesquelles il voyait une série de visages neutres de sa propre race ou d’une autre race associés à des extraits musicaux joyeux ou tristes.
- Propre race + condition de musique joyeuse (own-happy)
- Propre race + musique triste (own-sad)
- Autre race + musique heureuse (autre-heureux)
- Autre race + musique triste (autre-mal)
Dans le cadre de l’expérience “own-happy”, les enfants ont regardé six vidéos de visages asiatiques associées de manière séquentielle à six extraits musicaux joyeux. Dans l’autre condition, les enfants ont regardé six vidéos de visages africains associés séquentiellement à des extraits musicaux joyeux. En général, les conditions étaient identiques sur le plan procédural, à l’exception de la composition du visage et de la musique. Les mouvements oculaires des enfants ont été enregistrés à l’aide d’un système de suivi des yeux.
Votre objectif est de déterminer comment la race du visage et l’émotion de la musique, ainsi que leur interaction, influencent le comportement des nourrissons en matière de regard.
Vos variables indépendantes :
- Visage.race(chinois/africain)
- Musique.émotion(joyeux/triste)
Vos variables dépendantes :
- First.Face.Looking.Time : il s’agit du temps de regard sur la vidéo du premier visage dans les quatre conditions.
- Total.Looking.Time : Somme des temps de regard de chaque enfant sur les cinq visages suivants pour créer une mesure de leur temps de regard total sur les cinq visages après.
Commençons par charger les bibliothèques nécessaires et le jeu de données “BabyData”. Pour ce faire, téléchargez le fichier “infant_eye_tracking_study.csv” et exécutez le code suivant. N’oubliez pas de remplacer ‘path_to_your_downloaded_file’ par le chemin réel du jeu de données sur votre système.
Remarque : les cases grisées contiennent le code R, le signe “#” indiquant un commentaire qui ne s’exécutera pas dans RStudio.
BabyData <- read.csv('path_to_your_downloaded_file/infant_eye_tracking_study.csv')
library(rstatix) #for performing basic statistical tests
library(dplyr) #for sorting data
library(tidyr) #for data sorting and structure
library(ggplot2) #for visualizing your data
library(readr)
library(ggpubr)
library(gridExtra)
Fichiers à télécharger :
- infant_eye_tracking_study.csv
Veuillez compléter les exercices ci-joints au mieux de vos capacités.
An interactive H5P element has been excluded from this version of the text. You can view it online here:
https://ecampusontario.pressbooks.pub/rspnc/?p=152#h5p-3
Solutions
Exercice 1 – Préparation et exploration des données
Remarque : les cases grisées contiennent le code R, tandis que les cases blanches affichent la sortie du code, telle qu’elle apparaît dans RStudio.
Le signe “#” indique un commentaire qui ne sera pas exécuté dans RStudio.
1. Affichez les premières lignes pour comprendre votre jeu de données.
summary(BabyData) # Viewing the summary of the dataset to check for inconsistencies
## Age.in.Days Condition Face.Race Music.Emotion Age.Group
## 1 93 Other-Race Happy Music African happy 3
## 2 98 Other-Race Happy Music African happy 3
## 3 93 Other-Race Happy Music African happy 3
## 4 93 Other-Race Happy Music African happy 3
## 5 93 Other-Race Happy Music African happy 3
## 6 100 Other-Race Happy Music African happy 3
## Total.Looking.Time First.Face.Looking.Time Participant.ID
## 1 44.035 8.273 HJOGM7704U
## 2 18.324 6.938 JHSEG5414N
## 3 24.600 4.225 OCQFX4970K
## 4 12.919 7.537 KLDOF5559R
## 5 12.755 4.230 HHPGJ9661Y
## 6 38.777 9.351 NVCPX9518V
2. Utilisez relocate() pour réorganiser vos colonnes de manière à ce que la colonne “Participant.ID” apparaisse comme la première colonne de votre ensemble de données.
BabyData <- BabyData %>% relocate(Participant.ID, .before = Age.in.Days)
3. Vérifiez que vos données ne comportent pas de valeurs manquantes. Supprimez de l’ensemble de données toutes les lignes contenant des valeurs manquantes ou NA.
sum(is.na(BabyData)) # Checking for missing values in the dataset
BabyData <- BabyData[!is.na(BabyData$First.Face.Looking.Time), ]
## Participant.ID Age.in.Days Condition Face.Race
## Length:193 Min. : 79.0 Length:193 Length:193
## Class :character 1st Qu.:127.0 Class :character Class :character
## Mode :character Median :185.0 Mode :character Mode :character
## Mean :189.3
## 3rd Qu.:246.0
## Max. :316.0
##
## Music.Emotion Age.Group Total.Looking.Time First.Face.Looking.Time
## Length:193 Min. :3.000 Min. : 1.654 Min. : 0.160
## Class :character 1st Qu.:3.000 1st Qu.:20.671 1st Qu.: 5.309
## Mode :character Median :6.000 Median :30.381 Median : 7.495
## Mean :6.093 Mean :29.196 Mean : 7.041
## 3rd Qu.:9.000 3rd Qu.:38.196 3rd Qu.: 9.185
## Max. :9.000 Max. :50.000 Max. :11.823
## NA's :3
4. Vérifiez à nouveau que vos données ne comportent pas de valeurs manquantes et que les données sont cohérentes.
sum(is.na(BabyData)) # Checking for missing values in the dataset
summary(BabyData) # Viewing the summary of the dataset to check for inconsistencies
## Participant.ID Age.in.Days Condition Face.Race
## Length:193 Min. : 79.0 Length:193 Length:193
## Class :character 1st Qu.:127.0 Class :character Class :character
## Mode :character Median :185.0 Mode :character Mode :character
## Mean :189.3
## 3rd Qu.:246.0
## Max. :316.0
##
## Music.Emotion Age.Group Total.Looking.Time First.Face.Looking.Time
## Length:193 Min. :3.000 Min. : 1.654 Min. : 0.160
## Class :character 1st Qu.:3.000 1st Qu.:20.671 1st Qu.: 5.309
## Mode :character Median :6.000 Median :30.381 Median : 7.495
## Mean :6.093 Mean :29.196 Mean : 7.041
## 3rd Qu.:9.000 3rd Qu.:38.196 3rd Qu.: 9.185
## Max. :9.000 Max. :50.000 Max. :11.823
## NA's :0
5. Vérifiez la structure et assurez-vous que vos colonnes factorielles (Music.Emotion, Face.Race et Condition) sont correctement configurées.
## 'data.frame': 190 obs. of 8 variables:
## $ Participant.ID : chr "HJOGM7704U" "JHSEG5414N" "OCQFX4970K" "KLDOF5559R" ...
## $ Age.in.Days : int 93 98 93 93 93 100 93 91 98 100 ...
## $ Condition : chr "Other-Race Happy Music" "Other-Race Happy Music" "Other-Race Happy Music" "Other-Race Happy Music" ...
## $ Face.Race : chr "African" "African" "African" "African" ...
## $ Music.Emotion : chr "happy" "happy" "happy" "happy" ...
## $ Age.Group : int 3 3 3 3 3 3 3 3 3 3 ...
## $ Total.Looking.Time : num 44 18.3 24.6 12.9 12.8 ...
## $ First.Face.Looking.Time: num 8.27 6.94 4.22 7.54 4.23 ...
BabyData$Face.Race <- as.factor(BabyData$Face.Race)
BabyData$Music.Emotion <- as.factor(BabyData$Music.Emotion)
BabyData$Condition <- as.factor(BabyData$Condition)
6. Vérifiez si votre modèle est équilibré ou déséquilibré.
table(BabyData$Age.Group, BabyData$Condition) #unbalanced design
##
## Other-Race Happy Music Other-Race Sad Music Own-Race Happy Music
## 3 16 12 12
## 6 15 19 19
## 9 14 17 17
##
## Own-Race Sad Music
## 3 17
## 6 15
## 9 17
Exercice 2 : Réalisation d’une analyse de régression linéaire multi-variable
7. Effectuez une régression linéaire multi-variable sur le temps de regard du premier visage en tant que variable prédite, avec le groupe, la race du visage et leurs interactions en tant que variables prédictives. Affichez le résultat.
lm_model1 <- lm(First.Face.Looking.Time ~ Age.Group*Face.Race, data = BabyData)
summary(lm_model1)
##
## Call:
## lm(formula = First.Face.Looking.Time ~ Age.Group * Face.Race,
## data = BabyData)
##
## Residuals:
## Min 1Q Median 3Q Max
## -7.4524 -1.4478 0.3645 2.0507 4.5670
##
## Coefficients:
## Estimate Std. Error t value Pr(>|t|)
## (Intercept) 5.50710 0.75573 7.287 8.75e-12 ***
## Age.Group 0.22815 0.11542 1.977 0.0496 *
## Face.RaceChinese -0.04233 1.05722 -0.040 0.9681
## Age.Group:Face.RaceChinese 0.05036 0.16071 0.313 0.7544
## ---
## Signif. codes: 0 '***' 0.001 '**' 0.01 '*' 0.05 '.' 0.1 ' ' 1
##
## Residual standard error: 2.658 on 186 degrees of freedom
## Multiple R-squared: 0.05411, Adjusted R-squared: 0.03885
## F-statistic: 3.546 on 3 and 186 DF, p-value: 0.01564
8. Effectuez une régression linéaire multivariable similaire à celle décrite dans la question précédente. La variable prédite doit être le temps total de recherche, avec comme prédicteurs le groupe d’âge, la race du visage, l’émotion musicale et leurs interactions.
lm_model2 <- model <- lm(Total.Looking.Time ~ Age.Group * Face.Race * Music.Emotion, data = BabyData)
summary(lm_model2)
##
## Call:
## lm(formula = Total.Looking.Time ~ Age.Group * Face.Race * Music.Emotion,
## data = BabyData)
##
## Residuals:
## Min 1Q Median 3Q Max
## -24.8431 -8.0316 -0.1786 8.2809 27.7472
##
## Coefficients:
## Estimate Std. Error t value
## (Intercept) 28.0472 4.5406 6.177
## Age.Group -0.5167 0.7144 -0.723
## Face.RaceChinese -11.5424 6.6960 -1.724
## Music.Emotionsad -15.2955 6.6960 -2.284
## Age.Group:Face.RaceChinese 3.0376 1.0229 2.970
## Age.Group:Music.Emotionsad 3.4057 1.0229 3.330
## Face.RaceChinese:Music.Emotionsad 26.8342 9.3820 2.860
## Age.Group:Face.RaceChinese:Music.Emotionsad -5.7421 1.4252 -4.029
## Pr(>|t|)
## (Intercept) 4.16e-09 ***
## Age.Group 0.47045
## Face.RaceChinese 0.08645 .
## Music.Emotionsad 0.02351 *
## Age.Group:Face.RaceChinese 0.00338 **
## Age.Group:Music.Emotionsad 0.00105 **
## Face.RaceChinese:Music.Emotionsad 0.00473 **
## Age.Group:Face.RaceChinese:Music.Emotionsad 8.22e-05 ***
## ---
## Signif. codes: 0 '***' 0.001 '**' 0.01 '*' 0.05 '.' 0.1 ' ' 1
##
## Residual standard error: 11.72 on 182 degrees of freedom
## Multiple R-squared: 0.1742, Adjusted R-squared: 0.1425
## F-statistic: 5.486 on 7 and 182 DF, p-value: 9.76e-06
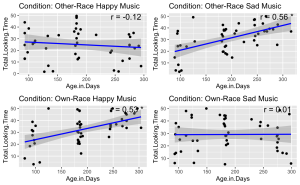
9. Compte tenu de l’interaction significative à trois voies, effectuer des analyses de corrélation de Pearson pour examiner la relation linéaire entre le temps total passé à regarder un visage et l’âge du participant en jours dans chaque condition.
- (a) Commence par identifier toutes les conditions uniques présentes dans l’ensemble de données.
- (b) Effectue une analyse de corrélation de Pearson entre le groupe d’âge et la durée totale d’observation pour chaque condition unique.
- (c) Enregistre et imprime les résultats de la corrélation, y compris les coefficients de corrélation et les valeurs p, pour chaque condition.
unique_conditions <- unique(BabyData$Condition) #Get unique conditions
correlation_results <- list() ## Initialize a list to store results
# Loop through each condition and perform Pearson correlation
for (condition in unique_conditions) {
# Subset data for the current condition
subset_data <- subset(BabyData, Condition == condition)
subset_data$Age.Group <- as.numeric(as.character(subset_data$Age.Group))
# Perform Pearson correlation
correlation_test <- cor.test(subset_data$Age.Group, subset_data$Total.Looking.Time, method = "pearson")
# Store the result
correlation_results[[condition]] <- correlation_test
}
# Print the results
correlation_results
## $`Other-Race Happy Music`
##
## Pearson's product-moment correlation
##
## data: subset_data$Age.Group and subset_data$Total.Looking.Time
## t = -0.64059, df = 43, p-value = 0.5252
## alternative hypothesis: true correlation is not equal to 0
## 95 percent confidence interval:
## -0.3799180 0.2020743
## sample estimates:
## cor
## -0.09722666
##
##
## $`Other-Race Sad Music`
##
## Pearson's product-moment correlation
##
## data: subset_data$Age.Group and subset_data$Total.Looking.Time
## t = 4.4535, df = 46, p-value = 5.356e-05
## alternative hypothesis: true correlation is not equal to 0
## 95 percent confidence interval:
## 0.3136678 0.7206311
## sample estimates:
## cor
## 0.5488839
##
##
## $`Own-Race Happy Music`
##
## Pearson's product-moment correlation
##
## data: subset_data$Age.Group and subset_data$Total.Looking.Time
## t = 3.8943, df = 46, p-value = 0.0003166
## alternative hypothesis: true correlation is not equal to 0
## 95 percent confidence interval:
## 0.2490419 0.6851408
## sample estimates:
## cor
## 0.4979416
##
##
## $`Own-Race Sad Music`
##
## Pearson's product-moment correlation
##
## data: subset_data$Age.Group and subset_data$Total.Looking.Time
## t = 0.25438, df = 47, p-value = 0.8003
## alternative hypothesis: true correlation is not equal to 0
## 95 percent confidence interval:
## -0.2466891 0.3149919
## sample estimates:
## cor
## 0.03707966
Exercice 3 : Visualisation des données
10. Visualisez la relation entre le temps total passé à regarder un visage et l’âge du participant en jours, en fonction des différentes conditions expérimentales. Chaque condition doit être représentée dans son propre panneau à l’intérieur d’une seule figure. En outre, pour chaque panneau :
- (a) Tracez le temps total passé par chaque enfant à regarder le visage en fonction de son âge en jours.
- (b) Ajoutez une ligne de régression linéaire bleue pour indiquer la tendance.
- (c) Affichez le coefficient de corrélation de Pearson que vous avez calculé à la question précédente dans le coin supérieur droit de chaque panneau. Pour l’affichage, arrondissez vos calculs à deux décimales.
- (d) Utilisez des panneaux différents pour chaque condition expérimentale et disposez-les en grille.
- (e) Veillez à ce qu’une corrélation significative (p < 0,05) soit indiquée par un astérisque.
# Get unique conditions
conditions <- unique(BabyData$Condition)
# Create a list to store plots
plot_list <- list()
# Loop through each condition and create a plot
for (condition in conditions) {
# Subset data for the condition
subset_data <- subset(BabyData, Condition == condition)
# Perform linear regression
fit <- lm(Total.Looking.Time ~ Age.in.Days, data = subset_data)
# Calculate Pearson correlation
cor_test <- cor.test(subset_data$Age.in.Days, subset_data$Total.Looking.Time)
# Create a scatter plot with regression line
p <- ggplot(subset_data, aes(x = Age.in.Days, y = Total.Looking.Time)) +
geom_point() +
geom_smooth(method = 'lm', color = 'blue') +
ggtitle(paste('Condition:', condition)) +
annotate("text", x = Inf, y = Inf, label = paste('r =', round(cor_test$estimate, 2), ifelse(cor_test$p.value < 0.05, "*", "")),
hjust = 1.1, vjust = 1.1, size = 5)
# Add plot to list
plot_list[[condition]] <- p
}
do.call(grid.arrange, c(plot_list, ncol = 2))
Exercice 4 : réalisation de tests T sur des échantillons indépendants
11. Analysez l’impact de la valence émotionnelle de la musique sur le temps de regard des visages de sa propre race et d’autres races dans différents groupes d’âge de nourrissons (3, 6 et 9 mois). Plus précisément, vous devez effectuer une série de tests t sur des échantillons indépendants.
- (a) En utilisant la colonne Age.Group, effectuez des tests t d’échantillons indépendants pour examiner les effets de la valence émotionnelle de la musique (Music. Emotion) sur le temps de regard (Total.Looking.Time) pour les visages de sa propre race et d’autres races (Face.Race) dans chaque groupe d’âge.
- (b) Assurez-vous que votre script tient compte des différentes combinaisons de groupes d’âge et de valences émotionnelles de la musique.
- (c) Stockez et affichez les résultats de ces tests t de manière organisée.
# Ensure Age.Group is treated as a factor
BabyData$Age.Group <- as.factor(BabyData$Age.Group)
# Perform t-tests for each combination of Age.Group, Music.Emotion, and Face.Race
results <- list()
for(age_group in levels(BabyData$Age.Group)) {
for(music_emotion in unique(BabyData$Music.Emotion)) {
# Filter data for specific age group and music emotion
subset_data <- BabyData %>%
filter(Age.Group == age_group, Music.Emotion == music_emotion)
# Perform the t-test comparing Total.Looking.Time for own- vs. other-race faces
t_test_result <- t.test(Total.Looking.Time ~ Face.Race, data = subset_data)
# Store the results
result_name <- paste(age_group, music_emotion, sep="_")
results[[result_name]] <- t_test_result
}
}
# Print results
print(results)
## $`3_happy`
##
## Welch Two Sample t-test
##
## data: Total.Looking.Time by Face.Race
## t = -0.3153, df = 22.294, p-value = 0.7555
## alternative hypothesis: true difference in means between group African and group Chinese is not equal to 0
## 95 percent confidence interval:
## -12.465591 9.173257
## sample estimates:
## mean in group African mean in group Chinese
## 22.76875 24.41492
##
##
## $`3_sad`
##
## Welch Two Sample t-test
##
## data: Total.Looking.Time by Face.Race
## t = -1.0492, df = 22.86, p-value = 0.3051
## alternative hypothesis: true difference in means between group African and group Chinese is not equal to 0
## 95 percent confidence interval:
## -17.297369 5.658457
## sample estimates:
## mean in group African mean in group Chinese
## 21.32725 27.14671
##
##
## $`6_happy`
##
## Welch Two Sample t-test
##
## data: Total.Looking.Time by Face.Race
## t = 0.43226, df = 27.324, p-value = 0.6689
## alternative hypothesis: true difference in means between group African and group Chinese is not equal to 0
## 95 percent confidence interval:
## -6.401791 9.821475
## sample estimates:
## mean in group African mean in group Chinese
## 32.90100 31.19116
##
##
## $`6_sad`
##
## Welch Two Sample t-test
##
## data: Total.Looking.Time by Face.Race
## t = -0.62019, df = 27.075, p-value = 0.5403
## alternative hypothesis: true difference in means between group African and group Chinese is not equal to 0
## 95 percent confidence interval:
## -9.635393 5.162123
## sample estimates:
## mean in group African mean in group Chinese
## 30.20163 32.43827
##
##
## $`9_happy`
##
## Welch Two Sample t-test
##
## data: Total.Looking.Time by Face.Race
## t = -6.0414, df = 21.29, p-value = 5.08e-06
## alternative hypothesis: true difference in means between group African and group Chinese is not equal to 0
## 95 percent confidence interval:
## -27.28467 -13.31931
## sample estimates:
## mean in group African mean in group Chinese
## 19.13607 39.43806
##
##
## $`9_sad`
##
## Welch Two Sample t-test
##
## data: Total.Looking.Time by Face.Race
## t = 3.0179, df = 26.642, p-value = 0.005546
## alternative hypothesis: true difference in means between group African and group Chinese is not equal to 0
## 95 percent confidence interval:
## 3.335708 17.533234
## sample estimates:
## mean in group African mean in group Chinese
## 38.68853 28.25406
Exercice 5 Création d’un diagramme à barres
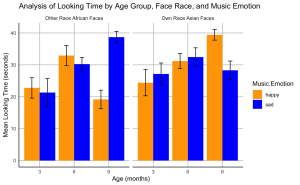
12. Créez un diagramme à barres pour visualiser les effets de la valence émotionnelle de la musique sur le temps de regard des nourrissons à différents âges pour les visages de leur propre race et ceux d’une autre race.
- (a) Le graphique doit montrer le temps total moyen passé à regarder les visages de sa propre race et ceux d’une autre race associés à une musique joyeuse ou triste pour chaque groupe d’âge.
- (b) Incluez les barres d’erreur standard dans votre graphique.
- (c) Organisez les barres de manière à ce que les barres représentant les visages de votre propre race soient regroupées et étiquetées “Visages asiatiques de votre propre race”, suivies des barres représentant les visages d’autres races regroupées et étiquetées “Visages africains d’autres races”.
- (d) La couleur des barres doit représenter l’émotion musicale : utilisez le bleu pour la musique triste et l’orange pour la musique joyeuse.
- (e) Inscrivez sur l’axe des x “Âge (mois)” et sur l’axe des y “Temps de regard moyen (secondes)”.
- (f) Intitulez votre graphique “Analyse du temps de regard par tranche d’âge, par race de visage et par émotion musicale”
- (g) Définissez le thème de votre graphique comme étant minimal. Assurez-vous que les lignes des axes x et y sont des lignes noires pleines.
- (h) Votre graphique ne doit pas afficher de lignes de grille mineures, mais uniquement des lignes de grille majeures.
# Calculate means and standard errors
data_summary <- BabyData %>%
group_by(Age.Group, Face.Race, Music.Emotion) %>%
summarize(Mean = mean(Total.Looking.Time),
SE = sd(Total.Looking.Time)/sqrt(n())) %>%
ungroup()
## `summarise()` has grouped output by 'Age.Group', 'Face.Race'. You can override
## using the `.groups` argument.
# Create the bar plot
ggplot(data_summary, aes(x = factor(Age.Group), y = Mean, fill = Music.Emotion)) +
geom_bar(stat = "identity", position = position_dodge()) +
geom_errorbar(aes(ymin = Mean - SE, ymax = Mean + SE),
position = position_dodge(0.9), width = 0.25) +
scale_fill_manual(values = c("happy" = "orange", "sad" = "blue")) +
facet_wrap(~ Face.Race, scales = "free_x", labeller = labeller(Face.Race = c(Chinese = "Own Race Asian Faces", African = "Other Race African Faces"))) +
labs(x = "Age (months)", y = "Mean Looking Time (seconds)", title = "Analysis of Looking Time by Age Group, Face Race, and Music Emotion") +
theme_minimal() +
theme(
panel.grid.minor = element_blank(),
panel.grid.major = element_line(color = "gray", size = 0.5, linetype = "solid"), # Major grid lines
axis.line = element_line(color = "black", size = 0.5) # Axis lines
)
P04: Laboratoire de perception et de sensorimotricité
Sevda Montakhaby Nodeh
Laboratoire de perception et de sensorimotricité
Bienvenue au laboratoire de perception et de sensorimotricité de l’université McMaster. En tant que psychologue cognitif en herbe, vous êtes sur le point d’entreprendre un voyage exploratoire sur l’effet de profondeur, un phénomène psychologique captivant qui suggère que les événements visuels se produisant à proximité (espace proche) sont traités plus efficacement que ceux qui sont plus éloignés (espace lointain). Cet effet offre une fenêtre unique sur l’architecture cognitive qui sous-tend nos expériences sensorielles, impliquant probablement l’implication du flux visuel dorsal, qui traite les relations spatiales et les mouvements dans l’espace proche, et le flux ventral, connu pour son rôle dans la reconnaissance d’informations visuelles détaillées.
Votre objectif est de déterminer si l’effet de profondeur est dépendant de la tâche, s’alignant strictement sur la dichotomie flux dorsal/flux ventral, ou s’il représente un avantage de traitement universel pour les stimuli dans l’espace proche à travers diverses tâches cognitives.
Votre parcours de recherche commence dans votre laboratoire. Imaginez le laboratoire comme une passerelle vers un monde tridimensionnel, où le concept de profondeur n’est pas seulement un sujet d’étude, mais aussi une expérience sensorielle vécue par vos participants ! Assis à l’intérieur d’une tente sombre, chaque participant tient un volant, son principal outil d’interaction et de saisie de réponses. Devant eux, un écran prend vie avec un environnement virtuel en 3D méticuleusement conçu pour tester les limites de la perception de la profondeur.
Le paysage virtuel auquel les participants sont confrontés est un modèle de simplicité et de complexité ; comme l’illustre la figure ci-dessous, devant les participants, un plan au sol s’étend dans la profondeur de l’écran, entrecoupé par deux séries de murs verticaux à des profondeurs variables – proches et éloignés. Les murs se trouvent de part et d’autre de l’axe central et se reflètent parfaitement sur la ligne médiane. Les textures du sol et des espaces réservés – une matrice de points aléatoires et un motif en damier, respectivement – conservent une densité constante. Ces indices visuels, ainsi que les gradients texturaux et la différence de taille rétinienne entre les objets proches et éloignés, agissent comme des repères subtils pour la perception de la profondeur.

De leur point de vue à la première personne, les participants sont invités à :
- Soit discriminer l’orientation d’une cible triangulaire rouge, soit localiser un carré à carreaux dans un environnement immersif en 3D.
- Les cibles peuvent apparaître dans des espaces proches ou éloignés, ce qui exige une discrimination et une localisation sensorielles poussées.
Grâce à cette expérience, vous ne vous contentez pas d’observer l’effet de profondeur ; vous le disséquerez, en découvrant les processus cognitifs qui permettent aux humains de naviguer dans la danse complexe de la profondeur dans notre vie quotidienne !
Commençons par charger les bibliothèques requises et le jeu de données. Pour ce faire, téléchargez le fichier “NearFarRep_Outlier.csv” et exécutez le code suivant.
Remarque : les cases grisées contiennent le code R, le signe “#” indiquant un commentaire qui ne s’exécutera pas dans RStudio.
# Loading the required
libraries library(tidyverse) # for data manipulation
library(rstatix) # for statistical analyses
library(emmeans) # for pairwise comparisons
library(afex) # for running anova using aov_ez and aov_car
library(kableExtra) # formatting html ANOVA tables
library(ggpubr) # for making plots
library(grid) # for plots
library(gridExtra) # for arranging multiple ggplots for extraction
library(lsmeans) # for pairwise comparisons
Lisez l’ensemble de données téléchargé “NearFarRep_Outlier.csv” en tant que “NearFarData”. N’oubliez pas de remplacer “path_to_your_downloaded_file” par le chemin réel de l’ensemble de données sur votre système.
NearFarData <- read.csv('path_to_your_downloaded_file/NearFarRep_Outlier.csv')
L’ensemble de données contient les temps de réponse des participants et comprend les colonnes suivantes :
- “Response” indique le type de tâche (discrimination ou localisation)
- “Con” indique la profondeur de la cible (proche ou lointaine)
- “TarRT” représente les temps de réponse de la cible.
Fichiers à télécharger :
- NearFarRep_Outlier.csv
Veuillez compléter les exercices ci-joints au mieux de vos capacités.
An interactive H5P element has been excluded from this version of the text. You can view it online here:
https://ecampusontario.pressbooks.pub/rspnc/?p=156#h5p-4
Solutions
Exercice 1 : préparer et explorer les données
Remarque : les cases grisées contiennent le code R, tandis que les cases blanches affichent la sortie du code, telle qu’elle apparaît dans RStudio.
Le signe “#” indique un commentaire qui ne sera pas exécuté dans RStudio.
1. Affichez les premières lignes pour comprendre votre ensemble de données. Affichez tous les noms de colonnes de l’ensemble de données.
head(NearFarData) #Displaying the first few rows
## X ID Response Con TarRT
## 1 1 10 Loc Near 0.6200754
## 2 2 10 Loc Near 0.2219719
## 3 3 1 Loc Near 0.2270377
## 4 4 9 Loc Near 0.5270686
## 5 5 25 Loc Near 0.2272455
## 6 6 18 Loc Near 0.2292785
## [1] "X" "ID" "Response" "Con" "TarRT"
2. Définissez “Response” et “Con” comme facteurs, puis vérifiez la structure de vos données pour vous assurer que vos facteurs et niveaux sont correctement définis.
NearFarData <- NearFarData %>%
convert_as_factor(Response, Con)
str(NearFarData)
## 'data.frame': 11154 obs. of 5 variables:
## $ X : int 1 2 3 4 5 6 7 8 9 10 ...
## $ ID : int 10 10 1 9 25 18 4 9 8 18 ...
## $ Response: Factor w/ 2 levels "Disc","Loc": 2 2 2 2 2 2 2 2 2 2 ...
## $ Con : Factor w/ 2 levels "Far","Near": 2 2 2 2 2 2 2 2 2 2 ...
## $ TarRT : num 0.62 0.222 0.227 0.527 0.227 ...
3. Effectuer des contrôles de base des données pour vérifier les valeurs manquantes et la cohérence des données.
sum(is.na(NearFarData)) # Checking for missing values in the dataset
4. Convertissez les valeurs de votre colonne de mesures dépendantes “TarRT” en secondes.
NearFarData$TarRT <- NearFarData$TarRT * 1000
Exercice 2 : Visualiser les données
- En utilisant le paquetage “dplyr”, écrivez un code R pour calculer le temps de réponse moyen et l’erreur standard de la moyenne (SERT) pour chaque combinaison de vos deux facteurs (Response et Con).
# Calculate means and standard errors for each combination of 'Response' and 'Con'
summary_df <- NearFarData %>%
group_by(Response, Con) %>%
summarise(
MeanRT = mean(TarRT),
SERT = sd(TarRT) / sqrt(n())
)
6. En utilisant le package “ggplot2”, créez un graphique linéaire avec des barres d’erreur pour la tâche de discrimination.
- (a) L’axe des x doit représenter la profondeur de la cible (Con) et être étiqueté “Profondeur de la cible”.
- (b) L’axe des y doit représenter le temps de réponse moyen (MeanRT) et être étiqueté “RT (ms)”
- (c) Les barres d’erreur doivent représenter l’erreur standard de la moyenne (SERT).
- (d) Assurez-vous que le type de ligne est solide.
- (e) Fixez la valeur minimale de l’axe des y à 630 et la valeur maximale à 660.
# Now, using ggplot to create the plot
Disc.plot <- ggplot(data = filter(summary_df, Response=="Disc"), aes(x = Con, y = MeanRT, group = Response)) +
geom_line(aes(linetype = "Discriminating")) + # Add a linetype aesthetic
geom_errorbar(aes(ymin = MeanRT - SERT, ymax = MeanRT + SERT), width = 0.1) +
geom_point(size = 3) +
theme_gray() +
labs(
x = "Target Depth",
y = "RT (ms)",
color = "Experiment",
linetype = "Experiment") +
scale_linetype_manual(values = "dashed") + # Set the linetype for "Disc" to dashed
ylim(630, 660) # Set the y-axis limits
7. De même, créez un graphique linéaire avec des barres d’erreur pour la tâche Localisation. Utilisez une ligne pointillée pour ce tracé, avec les exceptions suivantes :
- (a) Assurez-vous que le type de ligne est en pointillés.
- (b) Fixez la valeur minimale de l’axe des y à 370 et la valeur maximale à 410.
Loc.plot <- ggplot(data = filter(summary_df, Response=="Loc"), aes(x = Con, y = MeanRT, group = Response)) +
geom_line(aes(linetype = "Localizing")) + # Add a linetype aesthetic
geom_errorbar(aes(ymin = MeanRT - SERT, ymax = MeanRT + SERT), width = 0.1) +
geom_point(size = 3) +
theme_gray() +
labs(
x = "Target Depth",
y = "RT (ms)",
color = "Experiment",
linetype = "Experiment") +
scale_linetype_manual(values = "solid") + # Set the line type for "Disc" to dashed
ylim(370, 410) # Set the y-axis limits
8. Enfin, utilisez la fonction grid.arrange() du package “gridExtra” pour superposer les tracés des tâches de discrimination et de localisation.
grid.arrange(Disc.plot, Loc.plot, ncol = 1) # Stack the plots on top of each other
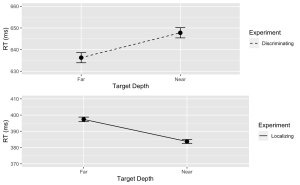
Exercice 3 : Analyse de la variance (ANOVA)
9. À l’aide de la fonction “anova_test”, effectuez une ANOVA à deux voies entre sujets pour étudier les effets de Con (condition) et de Response (type de tâche) sur les temps de réponse cibles (TarRT). Après avoir exécuté l’ANOVA, utilisez la fonction “get_anova_table” pour présenter les résultats.
anova <- anova_test(
data = NearFarData, dv = TarRT, wid = ID,
between = c(Con, Response), detailed = TRUE, effect.size = "pes")
## Warning: The 'wid' column contains duplicate ids across between-subjects
## variables. Automatic unique id will be created
get_anova_table(anova)
## ANOVA Table (type III tests)
##
## Effect SSn SSd DFn DFd F p p<.05
## 1 (Intercept) 2.972143e+09 111582983 1 11150 296993.268 0.00e+00 *
## 2 Con 3.185839e+03 111582983 1 11150 0.318 5.73e-01
## 3 Response 1.762985e+08 111582983 1 11150 17616.741 0.00e+00 *
## 4 Con:Response 4.390483e+05 111582983 1 11150 43.872 3.67e-11 *
## pes
## 1 9.64e-01
## 2 2.86e-05
## 3 6.12e-01
## 4 4.00e-03
Exercice 4: Analyse Post-Hoc
- Utilisez la fonction “lm” pour ajuster un modèle linéaire à vos données. Veillez à spécifier votre variable dépendante, vos variables indépendantes et vos termes d’interaction.
## Fitting a linear model to data
lm_model <- lm(TarRT ~ Con * Response, data = NearFarData)
- Utilisez la fonction “emmeans” pour obtenir les moyennes marginales estimées pour vos facteurs et leur interaction. Utilisez ensuite la fonction “pairs” pour effectuer des comparaisons par paire.
- (a) Réglez le paramètre adjust de la fonction test sur “Tukey” pour le test de différence significative honnête de Tukey, afin d’ajuster les comparaisons multiples et de contrôler le taux d’erreur de la famille.
# Get the estimated marginal means
emm <- emmeans(lm_model, specs = pairwise ~ Con * Response)
12. Imprimez et examinez les résultats de votre analyse post hoc. Les résultats fourniront une comparaison de chaque paire de niveaux de groupe, la différence estimée, l’erreur standard, la valeur t et la valeur p ajustée pour chaque comparaison.
# View the results
print(post_hoc_results)
## contrast estimate SE df t.ratio p.value
## Far Disc - Near Disc -11.5 2.70 11150 -4.250 0.0001
## Far Disc - Far Loc 238.9 2.67 11150 89.348 <.0001
## Far Disc - Near Loc 252.6 2.67 11150 94.581 <.0001
## Near Disc - Far Loc 250.4 2.69 11150 93.131 <.0001
## Near Disc - Near Loc 264.0 2.68 11150 98.341 <.0001
## Far Loc - Near Loc 13.6 2.66 11150 5.124 <.0001
##
## P value adjustment: tukey method for comparing a family of 4 estimates
P05 : Laboratoire d'éducation et de cognition
Sevda Montakhaby Nodeh
Laboratoire d’éducation et de cognition
Vous êtes chercheur au sein du laboratoire EdCog de l’Université McMaster. Le laboratoire mène une étude visant à comprendre les croyances des enseignants sur les capacités des étudiants dans les disciplines STIM (sciences, technologie, ingénierie et mathématiques). Cette étude est motivée par un nombre croissant de publications suggérant que les croyances des enseignants sur l’intelligence et la réussite – classées en croyances sur la brillance (l’idée que la réussite requiert un talent inné), croyances sur l’universalité (la croyance que la réussite est accessible à tous plutôt qu’à quelques privilégiés), et croyances sur l’état d’esprit (l’idée que l’intelligence et les compétences sont soit fixes, soit peuvent évoluer avec le temps) – jouent un rôle crucial dans les pratiques éducatives et les résultats des élèves. La compréhension de ces croyances est particulièrement importante dans les domaines des STIM, où les perceptions du talent inné par rapport aux compétences acquises peuvent influencer de manière significative les approches pédagogiques et l’engagement des étudiants.
Conception expérimentale :
L’enquête a été distribuée via LimeSurvey à des instructeurs des facultés de Sciences, de Sciences de la Santé et d’Ingénierie. Les participants ont été interrogés à travers une série de questions à l’échelle de Likert (allant de tout à fait en désaccord à tout à fait d’accord) visant à évaluer leurs croyances dans chacun des trois domaines. Des questions supplémentaires sur les données démographiques et le contexte ont été incluses pour contrôler des variables telles que les années d’expérience en enseignement, le domaine de spécialisation et le niveau d’éducation.
- Croyance en la Brilliance : La croyance que seuls ceux qui ont un talent brut et inné peuvent réussir dans leur domaine.
- Croyance en l’Universalité : La croyance que le succès est réalisable pour tout le monde, à condition que les bons efforts et stratégies soient employés.
- Croyances sur l’État d’Esprit : Les points de vue des instructeurs sur la nature de l’intelligence et des compétences – qu’ils soient des traits fixes ou qu’ils puissent être développés au fil du temps.
Le fichier de données d’échantillon (“EdCogData.xlsx”) pour cet exercice est structuré comme suit :
- ID : Un identifiant unique pour chaque répondant.
- Brilliance1 à Brilliance5 : Réponses aux déclarations mesurant la croyance en la brilliance comme une exigence pour le succès.
- Un score plus élevé dans ces colonnes indique une croyance que la brilliance est une exigence pour le succès.
- MindsetGrowth1 à MindsetGrowth5 : Réponses aux questions visant à évaluer la croyance en un état d’esprit de croissance, suggérant que l’intelligence et les capacités peuvent se développer avec le temps.
- Un score plus élevé dans ces colonnes indique un fort état d’esprit de croissance.
- Nonuniversality1 à Nonuniversality5 : Réponses aux déclarations mesurant les croyances contraires à l’universalité, signifiant que tout le monde ne peut pas réussir (c’est-à-dire que le succès n’est pas universel).
- Un score plus élevé dans ces colonnes indique un état d’esprit non universel face au succès.
- Universality1 à Universality5 : Réponses aux déclarations mesurant la croyance en l’universalité, ou l’idée que le succès est réalisable par n’importe qui avec un effort suffisant.
- Un score plus élevé dans ces colonnes indique une croyance qu’avec assez d’effort le succès est réalisable (c’est-à-dire que le succès est universel)
- MindsetFixed1 à MindsetFixed5 : Réponses aux questions visant à évaluer la croyance en un état d’esprit fixe concernant l’intelligence et les capacités. Un état d’esprit fixe croit que l’intelligence, les talents et les capacités sont des traits fixes. Ils pensent que ces traits sont innés et ne peuvent pas être significativement développés ou améliorés par l’effort ou l’éducation.
- Un score plus élevé dans ces colonnes indique un fort état d’esprit fixe.
Commencer : Charger les progiciels , définir le répertoire de travail et charger l’ensemble de données
Commençons par exécuter le code suivant dans RStudio pour charger les bibliothèques requises. Assurez-vous de lire les commentaires intégrés tout au long du code pour comprendre ce que chaque ligne de code fait.
Note : Les boîtes ombragées contiennent le code R, avec le signe “#” indiquant un commentaire qui ne s’exécutera pas dans RStudio.
# Here we create a list called "my_packages" with all of our required libraries
my_packages <- c("tidyverse", "readxl", "xlsx", "dplyr", "ggplot2")
# Checking and extracting packages that are not already installed
not_installed <- my_packages[!(my_packages %in% installed.packages()[ , "Package"])]
# Install packages that are not already installed
if(length(not_installed)) install.packages(not_installed)
# Loading the required libraries
library(tidyverse) # for data manipulation
library(dplyr) # for data manipulation
library(readxl) # to read excel files
library(xlsx) # to create excel files
library(ggplot2) # for making plots
Assurez-vous d’avoir téléchargé l’ensemble de données requis (“EdCogData.xlsx”) pour cet exercice. Définissez le répertoire de travail de votre session R actuelle dans le dossier contenant l’ensemble de données téléchargé. Vous pouvez le faire manuellement dans le studio R en cliquant sur l’onglet “Session” en haut de l’écran, puis en cliquant sur “Set Working Directory”.
Si le fichier de données téléchargé et votre session R se trouvent dans le même dossier, vous pouvez choisir de définir votre répertoire de travail sur “l’emplacement du fichier source” (l’emplacement où votre session R actuelle est sauvegardée). S’ils se trouvent dans des dossiers différents, cliquez sur l’option “choisir un répertoire” et recherchez l’emplacement du jeu de données téléchargé.
Vous pouvez également effectuer cette opération en exécutant le code suivant :
Une fois que vous avez défini votre répertoire de travail manuellement ou par code, la console ci-dessous affiche le répertoire complet de votre dossier en sortie.
Lisez l’ensemble de données téléchargé en tant que “edcogData” et effectuez les exercices qui l’accompagnent au mieux de vos capacités.
# Read xlsx file
edcog = read_excel("EdCogData.xlsx")
Fichiers à télécharger :
- EdCogData.xlsx
An interactive H5P element has been excluded from this version of the text. You can view it online here:
https://ecampusontario.pressbooks.pub/rspnc/?p=161#h5p-5
Solutions
Exercice 1 : Préparation et exploration des données
Note : Les boîtes ombragées contiennent le code R, tandis que les boîtes blanches affichent la sortie du code, telle qu’elle apparaît dans RStudio. Le signe “#” indique un commentaire qui ne s’exécutera pas dans RStudio.
Chargez l’ensemble de données dans RStudio et inspectez sa structure.
- Combien de lignes et de colonnes y a-t-il dans l’ensemble de données ?
- Quels sont les noms des colonnes ?
head(edcogData) # View the first few rows of the dataset
ncol(edcogData) #Q1
#[1] "ID" "Brilliance1" "Brilliance2" "Brilliance3" "Brilliance4"
#[6] "Brilliance5" "MindsetGrowth1" "MindsetGrowth2" "MindsetGrowth3" "MindsetGrowth4"
#[11] "MindsetGrowth5" "MindsetFixed1" "MindsetFixed2" "MindsetFixed3" "MindsetFixed4"
#[16] "MindsetFixed5" "Nonuniversality1" "Nonuniversality2" "Nonuniversality3" "Nonuniversality4"
#[21] "Nonuniversality5" "Universality1" "Universality2" "Universality3" "Universality4"
#[26] "Universality5"
Exercice 2 : Prétraitement des données
Préparez les données pour l’analyse en vous assurant qu’elles sont dans le bon format.
1. Y a-t-il des valeurs manquantes dans l’ensemble de données ?
Exercice 3 : Agrégation des scores
1. Créez des scores agrégés pour chaque dimension (Brilliance, Fixed, Growth, Nonuniversal, Universal).
edcogData$Brilliance <- rowMeans(edcogData[,c("Brilliance1", "Brilliance2", "Brilliance3", "Brilliance4", "Brilliance5")])
edcogData$Growth <- rowMeans(edcogData[,c("MindsetGrowth1", "MindsetGrowth2", "MindsetGrowth3", "MindsetGrowth4", "MindsetGrowth5")])
edcogData$Fixed <- rowMeans(edcogData[,c("MindsetFixed1", "MindsetFixed2", "MindsetFixed3", "MindsetFixed4", "MindsetFixed5")])
edcogData$Universal <- rowMeans(edcogData[,c("Universality1", "Universality2", "Universality3", "Universality4", "Universality5")])
edcogData$Nonuniversal <- rowMeans(edcogData[,c("Nonuniversality1", "Nonuniversality2", "Nonuniversality3", "Nonuniversality4", "Nonuniversality5")])
2. Créez un nouveau dataframe nommé “edcog.agg.wide” qui ne contient que la colonne ID et les colonnes de scores agrégés de “edcogData”.
edcog.agg.wide <- edcogData %>% select(ID, Brilliance, Fixed, Growth, Nonuniversal, Universal)
3. Convertissez “edcog.agg.wide” d’un format large à un format long nommé “edcog.agg.long”, avec les colonnes suivantes :
- ID
- Dimension (avec des valeurs de Brilliance, Fixed, Growth, Universal, et Nonuniversal)
- AggregateScore
edcog.agg.long <- edcog.agg.wide %>%
select(ID, Brilliance, Fixed, Growth, Nonuniversal, Universal) %>%
pivot_longer(
cols = -ID, # Select all columns except for ID
names_to = "Dimension",
values_to = "AggregateScore" )
Exercice 4 : Création de graphiques
1. Créez un boxplot pour visualiser la distribution des scores agrégés à travers différentes dimensions (Brilliance, Fixed, Growth, Nonuniversal, Universal) à partir des données de l’enquête avec les spécifications suivantes :
- L’axe des x doit représenter différentes ‘Dimensions’ de croyances.
- L’axe des y doit représenter le ‘Score’ sur une échelle de 1 à 5.
- Chaque ‘Dimension’ doit avoir une couleur différente pour sa boîte.
- Définissez l’étiquette de l’axe des y à “Réponse (sur une échelle de 1 à 5)” et l’étiquette de l’axe des x à “Dimension”.
- Utilisez un thème minimal et supprimez la légende. Indice :
- Utilisez “edcog.agg.long”
ggplot(edcog.agg.long, aes(x = Dimension, y = AggregateScore, fill = Dimension)) +
geom_boxplot() +
scale_fill_manual(values = c("Brilliance" = "pink", "Fixed" = "yellow",
"Growth" = "green", "Nonuniversal" = "blue",
"Universal" = "purple")) +
labs(y = "Response (on a 1 to 5 scale)", x = "Dimension") +
theme_minimal() +
theme(legend.position = "none") # Hide the legend since color coding is evident
2. Générez un graphique de violon pour visualiser la distribution des scores agrégés pour différentes dimensions (Brilliance, Fixed, Growth, Nonuniversal, Universal) à partir des données de l’enquête avec les spécifications suivantes :
- L’axe des x doit représenter différentes ‘Dimensions’ de croyances.
- L’axe des y doit représenter le ‘Score’ sur une échelle de 1 à 5.
- Chaque ‘Dimension’ doit avoir une couleur distincte.
- Étiquetez les axes de manière appropriée.
- Appliquez un thème minimaliste et envisagez de supprimer la légende si elle n’est pas nécessaire.
ggplot(edcog.agg.long, aes(x = Dimension, y = AggregateScore, fill = Dimension)) +
geom_violin(trim = FALSE) +
scale_fill_manual(values = c("Brilliance" = "pink", "Fixed" = "yellow",
"Growth" = "green", "Nonuniversal" = "blue",
"Universal" = "purple")) +
labs(y = "Response (on a 1 to 5 scale)", x = "Dimension") +
theme_minimal() + theme(legend.position = "none")
3. Améliorez le graphique de violon en superposant des points de données individuels pour montrer la distribution des données brutes en parallèle avec les estimations de densité agrégées.
ggplot(edcog.agg.long, aes(x = Dimension, y = AggregateScore, fill = Dimension)) +
geom_violin(trim = FALSE) +
geom_jitter(width = 0.1, size = 2, alpha = 0.5) + # Adjust 'width' for jittering, 'size' for point size, and 'alpha' for transparency
scale_fill_manual(values = c("Brilliance" = "pink", "Fixed" = "yellow",
"Growth" = "green", "Nonuniversal" = "blue",
"Universal" = "purple")) +
labs(y = "Response (on a 1 to 5 scale)", x = "Dimension") +
theme_minimal() +
theme(legend.position = "none")
Assurez-vous d’avoir l’ensemble de données requis (“EdCogData.xlsx”) pour cet exercice téléchargé. Définissez le répertoire de travail de votre session R actuelle sur le dossier contenant l’ensemble de données téléchargé. Vous pouvez le faire manuellement dans R studio en cliquant sur l’onglet “Session” en haut de l’écran, puis en cliquant sur “Définir le répertoire de travail”.
Si le fichier de l’ensemble de données téléchargé et votre session R sont dans le même fichier, vous pouvez choisir l’option de définir votre répertoire de travail sur “l’emplacement du fichier source” (l’emplacement où votre session R actuelle est enregistrée). S’ils sont dans des dossiers différents, cliquez sur l’option “choisir le répertoire” et recherchez l’emplacement de l’ensemble de données téléchargé.
Vous pouvez également le faire en exécutant le code suivant :
Une fois que vous avez défini votre répertoire de travail soit manuellement, soit par code, dans la console ci-dessous, vous verrez le répertoire complet de votre dossier en sortie.
Lisez l’ensemble de données téléchargé en tant que “edcogData” et complétez les exercices d’accompagnement au mieux de vos capacités.
P06: Laboratoire de la psychologie évolutionniste de la dépression
Carmen Tu
Rumination liée au deuil
Vous êtes un chercheur clinique à l’Hôpital Général de Hamilton, et votre laboratoire étudie comment les gens vivent le deuil et font face à la perte d’un être cher. Plus précisément, certaines personnes ruminent lorsqu’elles sont en deuil, et votre laboratoire est donc intéressé à comprendre les différentes façons dont cette rumination liée au deuil se manifeste chez les gens. Andrews et al. (2021) ont récemment développé le Questionnaire d’Analyse de la Rumination en Deuil (BARQ) pour évaluer deux dimensions de la rumination : 1) la cause de la perte (c’est-à-dire, l’analyse des causes profondes – RCA) et 2) comment une personne réinvestit son temps de manière significative après la perte (c’est-à-dire, l’analyse du réinvestissement – RIA).
Votre laboratoire se pose les questions suivantes :
- Les femmes en deuil ruminent-elles plus que les hommes en deuil ?
- Les gens ruminent-ils plus lorsque la mort de l’être cher est traumatisante ?
- La rumination liée au deuil varie-t-elle en fonction du type de relation avec le défunt ?
- La rumination dépend-elle de l’âge du défunt ?
- La rumination dépend-elle de l’âge du participant ?
- La rumination dépend-elle du temps écoulé depuis la mort de l’être cher ?
- Quelle dimension du BARQ est la plus associée à la dépression ?
Comme Andrews et al. (2021), votre laboratoire décide de recueillir les informations suivantes à partir d’un questionnaire distribué à 50 répondants :
- L’âge du défunt au moment de la mort
- Le temps écoulé depuis le moment de la mort
- L’âge actuel du répondant lors de la complétion du questionnaire
- Le sexe du répondant (homme, femme, ou autre)
- La relation du défunt avec le répondant (c’est-à-dire, enfant, parent, conjoint, ou autre)
- Si la mort a été traumatisante (oui ou non)
- Le nombre moyen d’heures de sommeil par nuit après la mort (c’est-à-dire, moins de 3 heures, 4-5 heures, 6-8 heures, plus de 9 heures)
- Si le répondant a été prescrit des médicaments psychiatriques
- Si le répondant a été prescrit des médicaments psychiatriques, cela incluait-il un antidépresseur ?
- Réponses à 7 items sur le BARQ. Quatre items forment le facteur latent RCA, tandis que trois items forment le facteur latent RIA. Les répondants ont évalué chacun des sept items sur une échelle de Likert à 4 points (1 = “Jamais”, 2 = “Parfois”, 3 = “Souvent”, 4 = “Tout le temps”).
Pour répondre aux questions du laboratoire, veuillez exécuter les analyses suivantes.
- Chargez les données. Les données peuvent être trouvées [ICI]. Exécutez des statistiques descriptives sur les traits démographiques suivants de l’échantillon.
- Quel est l’âge moyen du défunt au moment de la mort dans cet échantillon ?
- Quel est le temps moyen écoulé depuis le moment de la mort dans cet échantillon ?
An interactive H5P element has been excluded from this version of the text. You can view it online here:
https://ecampusontario.pressbooks.pub/rspnc/?p=300#h5p-107
-
- Quelle proportion des répondants étaient des femmes ?
- Quelle proportion des répondants ont perdu un enfant ?
- Quelle proportion des décès étaient traumatisants ?
- Quelle proportion des répondants ont été prescrits des médicaments antidépresseurs ?
An interactive H5P element has been excluded from this version of the text. You can view it online here:
https://ecampusontario.pressbooks.pub/rspnc/?p=300#h5p-108
2. Effectuez une analyse factorielle confirmatoire (CFA) sur les sept éléments du BARQ. Les éléments 1 à 4 devraient former le facteur latent RCA, et les éléments 5 à 7 devraient former le facteur latent RIA.
-
- Quelle est l’erreur quadratique moyenne d’approximation (RMSEA) de la CFA à deux facteurs ?
An interactive H5P element has been excluded from this version of the text. You can view it online here:
https://ecampusontario.pressbooks.pub/rspnc/?p=300#h5p-109
-
- Quel est l’indice de comparaison d’ajustement (CFI) et le résidu quadratique moyen standardisé (srmr) de la CFA à deux facteurs ? Utilisez CFI >= .95 et srmr <= .08 comme valeurs seuils.
An interactive H5P element has been excluded from this version of the text. You can view it online here:
https://ecampusontario.pressbooks.pub/rspnc/?p=300#h5p-110
3. Comparez les moyennes des facteurs latents RCA et RIA entre les groupes suivants. Quelles comparaisons présentent des différences statistiquement significatives dans les moyennes de RCA et RIA ?
-
- Les répondants qui prennent des médicaments antidépresseurs vs ceux qui n’en prennent pas
- Femmes vs hommes
- Les répondants dont l’être cher décédé a vécu une mort traumatisante vs ceux qui n’en ont pas vécu
- Les répondants qui ont perdu un enfant vs ceux qui n’en ont pas perdu
An interactive H5P element has been excluded from this version of the text. You can view it online here:
https://ecampusontario.pressbooks.pub/rspnc/?p=300#h5p-111
4. Les trois variables temporelles dans le questionnaire peuvent présenter une multilinéarité entre elles : l’âge du défunt, l’âge actuel du répondant et le temps écoulé depuis la mort. Par exemple, l’âge du défunt et l’âge du répondant peuvent être colinéaires, surtout si la relation du défunt au répondant est celle d’un enfant. Pour tester la multilinéarité, évaluez le facteur d’inflation de la variance (VIF) d’un modèle de régression qui inclut les trois variables temporelles comme prédicteurs pour RCA et RIA. Un VIF de 1 signifie qu’il n’y a pas de corrélation entre les variables prédicteurs, tandis qu’un VIF supérieur à 5 indique une forte corrélation.
An interactive H5P element has been excluded from this version of the text. You can view it online here:
https://ecampusontario.pressbooks.pub/rspnc/?p=300#h5p-112
5. Créez un nuage de points du score du facteur latent RCA du participant (axe des y) par rapport à l’âge de l’enfant décédé (axe des x).
An interactive H5P element has been excluded from this version of the text. You can view it online here:
https://ecampusontario.pressbooks.pub/rspnc/?p=300#h5p-113
Fichiers à télécharger :
- P06_dataset.csv
Références et lectures complémentaires :
Andrews, P. W., Altman, M., Sevcikova, M., & Cacciatore, J. (2021). An evolutionary approach to grief-related rumination: Construction and validation of the Bereavement Analytical Rumination Questionnaire. Evolution and Human Behavior, 42(5), 441-452.
P07 : Laboratoire de la psychologie narrative
Carmen Tu
Les caractéristiques du film peuvent-elles prédire la réception du public?
Les studios d’Hollywood et les producteurs de films sont vivement intéressés à déterminer quels types de scénarios résonneront avec le public et les critiques. Quelques scénaristes en herbe vous approchent, un chercheur en psychologie sociale spécialisé dans l’analyse de contenu qualitative, pour effectuer une analyse des intrigues de films afin de les aider à déterminer les types d’intrigues qui pourraient plaire aux téléspectateurs grand public. 150 films des cinq dernières années ont été sélectionnés au hasard et analysés pour leurs traits d’intrigue et de personnage. Vous décidez d’analyser les six caractéristiques narratives suivantes pour l’étude exploratoire : 1) le genre, 2) la forme de l’intrigue, 3) le type de but du protagoniste, 4) l’agence du protagoniste, 5) la coopérativité du protagoniste, et 6) l’assertivité du protagoniste. Vous êtes intéressé à voir comment ces caractéristiques se rapportent aux résultats suivants : 1) la note moyenne des critiques du film (en pourcentage) et 2) le bénéfice net du film (en dollars US).
Pour analyser ces six caractéristiques narratives, vous adoptez le schéma de Brown & Tu (2020) pour l’intrigue et le schéma de classification de Berry & Brown (2017) pour les personnages littéraires. Le schéma de codage pour les cinq caractéristiques narratives est le suivant :
-
Genre
| Étiquette |
Code |
| Drame |
1 |
| Comédie |
2 |
| Romance |
3 |
| Action |
4 |
| Horreur |
5 |
-
Forme du graphique
| Étiquette |
Code |
| Chute-Hausse |
1 |
| Chute-Hausse-Chute |
2 |
| Hausse-Chute |
3 |
| Hausse-Chute-Hausse |
4 |
-
But du protagoniste
| Étiquette |
Code |
| Efforcement |
1 |
| Faire Face |
2 |
-
Coopérativité du protagoniste
| Étiquette |
Code |
| Haute |
1 |
| Moyenne |
2 |
| Basse |
3 |
-
Assertion du protagoniste
| Étiquette |
Code |
| Haute |
1 |
| Moyenne |
2 |
| Basse |
3 |
Vous recrutez également un deuxième codeur afin de déterminer s’il y a une fiabilité inter-juges dans votre méthode de codage.
- Chargez les données. Les données peuvent être trouvées [ICI]. Exécutez des statistiques descriptives (par exemple, des mesures de fréquence, des mesures de tendance centrale, y compris la moyenne, la médiane et le mode, le cas échéant) sur chacune des six caractéristiques narratives, en utilisant les données de codage du Rater 1 (R1). Répondez aux questions suivantes :
-
- Quel est le genre de film le plus courant dans le corpus ?
- Quelle est la forme d’intrigue la plus courante dans le corpus ?
- Quel est le type de but du protagoniste le plus courant dans le corpus ?
- Quel est le type d’agence du protagoniste le plus courant dans le corpus ?
- Quel est le type de coopérativité du protagoniste le plus courant dans le corpus ?
- Quel est le type d’assertivité du protagoniste le plus courant dans le corpus ?
An interactive H5P element has been excluded from this version of the text. You can view it online here:
https://ecampusontario.pressbooks.pub/rspnc/?p=302#h5p-114
-
- Quelle est l’agence moyenne du protagoniste à travers les 150 films du corpus ?
- Quelle est la coopérativité moyenne du protagoniste à travers les 150 films du corpus ?
- Quelle est l’assertivité moyenne du protagoniste à travers les 150 films du corpus ?
An interactive H5P element has been excluded from this version of the text. You can view it online here:
https://ecampusontario.pressbooks.pub/rspnc/?p=302#h5p-115
- Répondez aux questions suivantes sur les deux variables de résultat
- Quels sont les cinq films ayant la note moyenne des critiques la plus élevée ?
- Quels sont les cinq films ayant le plus grand bénéfice net ?
- Quels sont les cinq films ayant le bénéfice net le plus faible ?
An interactive H5P element has been excluded from this version of the text. You can view it online here:
https://ecampusontario.pressbooks.pub/rspnc/?p=302#h5p-116
- Les six variables narratives sont un mélange de données nominales (genre, forme de l’intrigue, objectif du protagoniste) et de données ordinales (agence du protagoniste, coopérativité, assertivité). Le genre est la seule variable qui n’est pas codée par les évaluateurs. Quelles sont les relations entre les variables nominales ? Pour le déterminer, veuillez répondre aux questions suivantes :
- Quel genre de film a le pourcentage le plus élevé de formes d’intrigue de type 1 (chute-élévation) ?
- Quelle est la distribution en pourcentage des formes d’intrigue dans chaque genre ?
An interactive H5P element has been excluded from this version of the text. You can view it online here:
https://ecampusontario.pressbooks.pub/rspnc/?p=302#h5p-117
4. Visualisez ces distributions de formes d’intrigue dans chaque genre dans un graphique à barres groupées. Assurez-vous de bien étiqueter l’axe des y, l’axe des x et la légende.
-
- En guise de pratique, créez des graphiques à barres groupées entre n’importe quelle autre paire de variables afin de visualiser les interactions entre les variables.
An interactive H5P element has been excluded from this version of the text. You can view it online here:
https://ecampusontario.pressbooks.pub/rspnc/?p=302#h5p-118
5. Effectuez des analyses comparatives pour voir si l’une des six caractéristiques est liée à une autre. Comme les six caractéristiques ne sont pas normalement distribuées, utilisez des tests non paramétriques, tels que le Chi-Carré. Par exemple, la relation entre le genre et l’intrigue est-elle statistiquement significative ?
-
- L’hypothèse nulle est que la différence entre les données observées et les données attendues est due au hasard.
- Un résultat significatif du Chi-carré nous permettra de rejeter l’hypothèse nulle et de considérer une hypothèse alternative où la différence peut être due à la relation entre les deux variables.
An interactive H5P element has been excluded from this version of the text. You can view it online here:
https://ecampusontario.pressbooks.pub/rspnc/?p=302#h5p-119
6. Effectuez un test d’analyse de variance pour voir si l’une des cinq variables codées par l’évaluateur est liée à l’une des deux variables de résultat. Considérez si vous devez effectuer une ANOVA factorielle à 2 ou 3 voies. Quelles sont les hypothèses que vous devez considérer et tester avant de réaliser une ANOVA ?
An interactive H5P element has been excluded from this version of the text. You can view it online here:
https://ecampusontario.pressbooks.pub/rspnc/?p=302#h5p-120
7. Effectuez une analyse de regroupement pour voir si l’une des catégories des six variables de caractéristiques narratives se regroupe.
An interactive H5P element has been excluded from this version of the text. You can view it online here:
https://ecampusontario.pressbooks.pub/rspnc/?p=302#h5p-121
8. Déterminez la fiabilité inter-juges entre les deux codeurs pour chacune des cinq variables codées par l’évaluateur. Un score de Kappa de Cohen supérieur à 0,8 reflète un fort accord inter-juges.
An interactive H5P element has been excluded from this version of the text. You can view it online here:
https://ecampusontario.pressbooks.pub/rspnc/?p=302#h5p-122
Fichiers à télécharger :
- P07_dataset.csv
Références et lectures complémentaires :
Berry, M., & Brown, S. (2017). A classification scheme for literary characters. Psychological Thought, 10(2).
Brown, S., & Tu, C. (2020). The shapes of stories: A “resonator” model of plot structure. Frontiers of Narrative Studies, 6(2), 259-288.
P08: Laboratoire de la voix et de la personnalité
Carmen Tu
Intonation vocale et traits de personnalité
Un nouveau jeu de rôle en ligne massivement multijoueur (MMORPG) de science-fiction a été lancé, permettant aux joueurs de concevoir le son de la voix de leur avatar. Le jeu propose une échelle de hauteur glissante pour que les joueurs puissent sélectionner la hauteur fondamentale (f0) et les fréquences formantiques (pf) (toutes deux mesurées en hertz) de la voix de leur avatar. Au fur et à mesure que les joueurs s’engagent dans le monde du jeu et interagissent avec d’autres joueurs à travers leur avatar, des traits de personnalité spécifiques pour leur avatar émergent. Afin de déterminer si les avatars créés dans ce monde de jeu de science-fiction ressemblent aux modèles vocaux et de personnalité trouvés dans le monde réel, les concepteurs du jeu ont collaboré avec des psychologues pour enquêter sur cette comparaison. Les concepteurs du jeu ont été particulièrement inspirés par l’étude de Stern et al. (2021) qui a exploré comment la hauteur de la voix est liée à l’extraversion, la dominance et la sociosexualité autodéclarées chez les hommes et les femmes.
L’étude actuelle sur les jeux vidéo a recruté un échantillon de 475 joueurs du jeu, 200 hommes et 275 femmes, tous âgés de 20 à 45 ans. Dans l’étude de Stern et al. (2021), les participants liraient de courts passages de texte afin que les chercheurs puissent déterminer la hauteur fondamentale et les fréquences formant la voix des participants. Cependant, les concepteurs du jeu sont en mesure d’extraire les informations sur la voix d’un avatar à partir des sélections que le joueur a faites sur l’échelle de hauteur glissante lorsqu’il a créé son avatar. Les concepteurs du jeu ont demandé aux 475 joueurs de remplir chacun un questionnaire sur les traits de personnalité de leur avatar (évaluations de Likert sur une échelle de 1 à 5 du Big 5), la dominance (évaluations de Likert sur une échelle de 1 à 5 de la Liste des adjectifs interpersonnels, et la sociosexualité (évaluations de Likert sur une échelle de 1 à 5 du SOI-R). Le score de sociosexualité est la moyenne des évaluations pour les trois dimensions de la sociosexualité : comportement, attitude et désir. Les concepteurs du jeu ont déjà calculé l’alpha de Cronbach pour les scores bruts des échelles et ont constaté qu’il y avait une bonne fiabilité. Comme certains MMORPG peuvent offrir la possibilité aux joueurs d’explorer et d’exprimer leur sexualité à travers leurs avatars, comme le nouveau MMORPG de science-fiction actuel qui est l’objet de ce scénario, les concepteurs du jeu sont curieux de savoir s’il existe une relation entre ces traits autodéclarés et le type de voix que le joueur a conçu pour son avatar.
Pour aider les concepteurs du jeu à enquêter sur cette question, veuillez répondre aux questions suivantes :
Chargez les données. Les données peuvent être trouvées dans voiceData.
An interactive H5P element has been excluded from this version of the text. You can view it online here:
https://ecampusontario.pressbooks.pub/rspnc/?p=304#h5p-123
- Créez des nuages de points qui tracent f0 et pf par rapport aux neuf traits de personnalité : névrosisme, extraversion, ouverture, amabilité, conscience, dominance, et comportement sociosexuel, attitude, et désir. Codez les points de données par couleur en fonction du sexe.
- Appliquez un lissage par spline de régression à plaque mince sur les nuages de points pour diagnostiquer visuellement la non-linéarité. Voyez-vous des interactions possibles, des relations linéaires ou non linéaires dans les comparaisons bivariées ?
An interactive H5P element has been excluded from this version of the text. You can view it online here:
https://ecampusontario.pressbooks.pub/rspnc/?p=304#h5p-124
2. Créez des graphiques en violon comparant le sexe avec f0, pf, et les neuf variables de traits de personnalité. Inspectez visuellement les graphiques en violon et répondez aux questions suivantes:
-
- Quelle est la moyenne de f0 pour les femmes et les hommes ?
- Quelle est la moyenne de pf pour les femmes et les hommes ?
- Quelle est la moyenne des évaluations de Likert pour l’extraversion pour les femmes et les hommes ?
- Quelle est la moyenne des évaluations de Likert pour l’amabilité pour les femmes et les hommes ?
- Quelle est la moyenne des évaluations de Likert pour la dominance pour les femmes et les hommes ?
- Quelle est la moyenne des évaluations de Likert pour le comportement sociosexuel pour les femmes et les hommes ?
- La forme du graphique en violon montre-t-elle que les données ont une distribution bimodale, uniforme ou normale ?
An interactive H5P element has been excluded from this version of the text. You can view it online here:
https://ecampusontario.pressbooks.pub/rspnc/?p=304#h5p-125
- Exécutez une analyse de régression linéaire pour répondre aux questions suivantes :
- Les participants dont les avatars ont une hauteur fondamentale plus basse déclarent-ils leurs avatars comme étant plus élevés en névrosisme ?
- Les participants dont les avatars ont une hauteur fondamentale plus basse déclarent-ils leurs avatars comme étant plus élevés en extraversion ?
- Les participants dont les avatars ont une hauteur fondamentale plus basse déclarent-ils leurs avatars comme étant plus élevés en dominance ?
- Les participants dont les avatars ont une hauteur fondamentale plus basse déclarent-ils leurs avatars comme étant plus élevés en comportement sociosexuel ?
An interactive H5P element has been excluded from this version of the text. You can view it online here:
https://ecampusontario.pressbooks.pub/rspnc/?p=304#h5p-126
Références et lectures complémentaires :
Stern, J., Schild, C., Jones, B. C., DeBruine, L. M., Hahn, A., Puts, D. A., … & Arslan, R. C. (2021). Do voices carry valid information about a speaker’s personality?. Journal of Research in Personality, 92, 104092.
P09 : Synchronie musicale de LIVELab
Carmen Tu
Synchronie musicale et coordination interpersonnelle
Vous êtes chercheur dans une académie de musique en Ontario. Vous souhaitez comprendre comment les musiciens d’un quatuor à cordes se coordonnent et se synchronisent entre eux. Pour ce faire, vous recrutez deux groupes de musiciens pour le laboratoire Large Interactive Virtual Environment (LIVELab, https://livelab.mcmaster.ca) à l’Université McMaster où les données comportementales en direct des musiciens peuvent être enregistrées. Plus précisément, le balancement du corps a été mesuré à l’aide d’un système de capture de mouvement optique infrarouge, qui nécessite que chaque musicien porte une casquette en feutre avec des marqueurs réfléchissants.
Les deux groupes de musiciens sont deux quatuors à cordes. Un quatuor à cordes se compose d’un premier violoniste (étiqueté M1), d’un deuxième violoniste (M2), d’un altiste (M3) et d’un violoncelliste (M4). Les deux quatuors à cordes ont interprété le même extrait musical de 2 minutes trois fois (c’est-à-dire trois essais). Un quatuor à cordes a interprété l’extrait dans la même pièce (la condition “vue”), tandis que l’autre quatuor à cordes a interprété dans une pièce où il y avait des séparateurs entre les musiciens pour les empêcher de se voir (la condition “pas de vue”).
Le balancement du corps a été mesuré comme le changement de position en millimètres dans la direction antéro-postérieure. Les données peuvent être trouvées [ICI]. Les mesures ont été prises à une fréquence de 8hz (c’est-à-dire 8 échantillons de temps par seconde). Un enregistrement de 2 minutes donnera donc 960 points de données d’échantillons de temps par musicien.
La synchronie musicale et la coordination interpersonnelle peuvent être analysées en comparant à quel point la série temporelle du balancement du corps d’un musicien est similaire à celle d’un autre musicien (c’est-à-dire une corrélation croisée entre deux séries temporelles) ainsi qu’à savoir si la série temporelle d’un musicien est capable de prédire la série temporelle d’un autre musicien (c’est-à-dire une analyse de causalité de Granger entre les séries temporelles), ce qui est également connu sous le nom de flux d’information.
En tant que chercheur de l’académie de musique, vous aimeriez comprendre les questions suivantes :
Est-ce que les quatuors à cordes dans les conditions de vue et de non-vue deviennent plus synchronisés à chaque essai successif ? Le quatuor à cordes dans la condition de soupir est-il plus synchronisé que le quatuor à cordes dans la condition de non-vue ? Est-ce que la série temporelle du premier violoniste “prévoit” la série temporelle des autres musiciens ? Pour répondre à la question ci-dessus, veuillez compléter les analyses statistiques suivantes :
- Tracez une série temporelle de graphiques en ligne pour chaque musicien dans chaque essai. Comparez visuellement les séries de lignes entre les quatuors dans la condition de vue vs la condition de non-vue.
An interactive H5P element has been excluded from this version of the text. You can view it online here:
https://ecampusontario.pressbooks.pub/rspnc/?p=307#h5p-127
2. Convertissez les données de la chronologie de chaque musicien en scores z.
An interactive H5P element has been excluded from this version of the text. You can view it online here:
https://ecampusontario.pressbooks.pub/rspnc/?p=307#h5p-128
3. Exécutez une analyse de corrélation croisée de fenêtre entre les paires suivantes de musiciens dans chaque essai et dans chaque condition (il y a six combinaisons de paires possibles) :
-
- M1 et M2
- M1 et M3
- M1 et M4
- M2 et M3
- M2 et M4
- M3 et M4
An interactive H5P element has been excluded from this version of the text. You can view it online here:
https://ecampusontario.pressbooks.pub/rspnc/?p=307#h5p-129
4. Effectuez une analyse de causalité de Granger entre les paires suivantes de musiciens dans chaque essai et chaque condition (il y a 12 combinaisons de paires possibles)
-
- M1 -> M2 et M2 <- M1
- M1 -> M3 et M3 <- M1
- M1 -> M4 et M4 <- M1
- M2 -> M3 et M3 <- M2
- M2 -> M4 et M4 <- M2
- M3 -> M4 et M4 <- M3
An interactive H5P element has been excluded from this version of the text. You can view it online here:
https://ecampusontario.pressbooks.pub/rspnc/?p=307#h5p-130
5. En utilisant la modélisation à effets mixtes linéaires, déterminez si les corrélations croisées ont augmenté au fil des essais. Si c’est le cas, cela peut indiquer que le balancement du corps entre les musiciens devient plus similaire à mesure qu’ils se familiarisent avec la pièce.
-
- Les variables de résultat sont la similarité de groupe (corrélations croisées) et le flux d’information (causalité de Granger)
- Les essais sont l’effet fixe (3 essais)
- Il y a un effet aléatoire de paire (6 paires pour la similarité de groupe et 12 paires pour le flux d’information)
Fichiers à télécharger :
- P09_dataset.csv
Références et lectures complémentaires :
Wood, E. A., Chang, A., Bosnyak, D., Klein, L., Baraku, E., Dotov, D., & Trainor, L. J. (2022). Creating a shared musical interpretation: Changes in coordination dynamics while learning unfamiliar music together. Annals of the New York Academy of Sciences, 1516(1), 106-113.
P10 : Laboratoire sur les perceptions sociales
Carmen Tu
Les vues essentialistes des enfants sur les groupes nationaux
Vous êtes un chercheur en perceptions sociales et vous êtes intéressé par la question de savoir s’il existe des différences dans la façon dont les enfants du pays A et les enfants du pays B perçoivent la nationalité. Vous avez été spécifiquement inspiré par l’étude de Siddiqui, Cimpian et Rutherford (2020) comparant le degré de perspectives essentialistes des groupes nationaux entre les enfants canadiens et américains. Pour enquêter sur l’existence de différences dans les vues essentialistes sur les groupes nationaux, vous avez recruté 50 enfants âgés de 5 à 8 ans du pays A et 50 enfants âgés de 4 à 9 ans du pays B. Vous demandez aux enfants de répondre à des questions relatives aux catégories suivantes d’essentialisme :
- Stabilité
- Si les enfants supposent que chaque groupe national a des “essences”, alors ils devraient également percevoir l’appartenance au groupe national comme étant “instable”. Exemple : On montre à un enfant une photo d’une fille qui est étiquetée comme citoyenne du pays A. On demande à l’enfant si la fille restera citoyenne du pays A même si elle déménage.
- Hérédité
- La nationalité est-elle héréditaire ? Exemple : on dit à un enfant qu’un couple du pays B a un bébé, et que le bébé est adopté par une famille d’un autre pays. Le bébé est-il toujours citoyen du pays A ?
- Potentiel inductif
- Les enfants perçoivent-ils les membres du même groupe comme ayant des traits similaires ? Exemple : On dit à un enfant qu’une fille du pays A aime les pommes, tandis qu’un garçon du pays B aime les oranges. On demande à l’enfant quel fruit un garçon du pays A aimerait.
- Intérieurs
- L’appartenance est-elle biologique ? Exemple : On demande aux enfants si une personne pourrait être identifiée comme étant du pays A en regardant ses “intérieurs”, comme les os de l’individu.
- Tradition
- Les enfants attribuent-ils les traditions nationales aux préférences communes des citoyens de ce pays (par exemple, le sirop d’érable est un condiment courant dans les repas canadiens parce qu’il est aimé par les Canadiens) ou aux facteurs environnementaux du pays (par exemple, le sirop d’érable est un condiment courant dans les repas canadiens parce que les érables sont courants) ?
- Signification
- Qu’est-ce que cela signifie d’être citoyen d’un certain pays ? Exemple : Les enfants attribuent-ils l’identité nationale à un trait de comportement, comme être gentil ? Ou les enfants attribuent-ils l’identité nationale à l’endroit où vit un individu ?
- Acquisition
- Comment les enfants supposent-ils qu’un individu peut devenir citoyen d’un pays ? Exemple : Les enfants supposent-ils qu’être gentil fait d’un individu un membre d’un groupe national ? Ou les enfants croient-ils que le fait de déménager dans ce pays permet à cet individu de devenir membre de ce groupe national ?
Les options de réponse étaient binaires pour chaque question, où une réponse recevra un score de 1, tandis qu’une autre réponse recevra un score de zéro. Les questions dans la même catégorie ont été moyennées pour donner un score d’essentialisme pour cette catégorie. Les scores qui sont plus proches de 1 indiquent un essentialisme élevé. Les réponses des enfants peuvent être trouvées ci-dessous. Pour comparer si les vues essentialistes diffèrent entre les enfants du pays A et du pays B, veuillez exécuter les analyses statistiques suivantes :
- Calculez la moyenne pour les éléments suivants :
- Âge du participant enfant dans le pays A
- Âge du participant enfant dans le pays B
- Score d’essentialisme pour les enfants du pays A pour chacune des sept catégories
- Score d’essentialisme pour les enfants du pays B pour chacune des sept catégories
An interactive H5P element has been excluded from this version of the text. You can view it online here:
https://ecampusontario.pressbooks.pub/rspnc/?p=309#h5p-131
2. Créez un graphique de dispersion pour montrer les scores d’essentialisme.
An interactive H5P element has been excluded from this version of the text. You can view it online here:
https://ecampusontario.pressbooks.pub/rspnc/?p=309#h5p-132
3. Exécutez des comparaisons de tests t par paires entre les scores d’essentialisme pour chacune des sept catégories entre le pays A et le pays B.
An interactive H5P element has been excluded from this version of the text. You can view it online here:
https://ecampusontario.pressbooks.pub/rspnc/?p=309#h5p-133
4. Comme il y a plusieurs comparaisons, le risque d’erreur de type 1 est augmenté. Exécutez une correction de Bonferroni pour prendre en compte cela.
An interactive H5P element has been excluded from this version of the text. You can view it online here:
https://ecampusontario.pressbooks.pub/rspnc/?p=309#h5p-134
5. Créez un modèle linéaire à effets mixtes en utilisant les catégories d’essentialisme comme prédicteur catégoriel et l’âge des enfants comme prédicteur continu.
An interactive H5P element has been excluded from this version of the text. You can view it online here:
https://ecampusontario.pressbooks.pub/rspnc/?p=309#h5p-135
Fichiers à télécharger :
- P10_dataset.csv
Références et lectures complémentaires :
Siddiqui, H., Cimpian, A., & Rutherford, M. D. (2020). Canadian children’s concepts of national groups: A comparison with children from the United States. Developmental psychology, 56(11), 2102.
Recherche en Neuroscience
II
N01 : L'Électroencéphalogramme
Matin Yousefabadi
Une analyse de l’électroencéphalographie avec R : un guide pédagogique
Une introduction à l’électroencéphalographie (EEG)
L’électroencéphalogramme, ou EEG, est une technique de neuro-imagerie non invasive qui mesure et enregistre l’activité électrique du cerveau. Elle consiste à placer des électrodes sur le cuir chevelu pour détecter les fluctuations de tension résultant du courant ionique dans les neurones du cerveau. L’EEG fournit une représentation en temps réel de l’activité cérébrale et est largement utilisée en neurosciences informatiques pour étudier les processus neuronaux et comprendre le fonctionnement du cerveau.
En neurosciences informatiques l’analyse des données EEG se fait à l’aide d’algorithmes et de modèles mathématiques avancés afin d’extraire des informations précieuses sur les processus cognitifs, la perception sensorielle et divers troubles neurologiques. Les chercheurs utilisent l’EEG pour étudier les schémas d’activité neuronale, la connectivité cérébrale et la dynamique temporelle du traitement de l’information. La polyvalence et la précision temporelle de l’EEG en font un outil précieux pour étudier les fonctions cérébrales et contribuer à notre compréhension de l’interaction complexe des neurones dans le cerveau humain.
Les bandes de fréquence de l’EEG
Les signaux EEG sont caractérisés par différentes bandes de fréquence qui reflètent l’activité neuronale sous-jacente. Les bandes de fréquences sont définies en fonction de la gamme de fréquences du signal EEG, et chaque bande est associée à un type spécifique d’activité cérébrale. Les bandes de fréquence sont les suivantes :
- Ondes delta (δ) (0,5-4 Hz) :
- Les ondes delta sont importantes pendant le sommeil profond et indiquent les oscillations les plus lentes du cerveau.
- Les anomalies de l’activité delta peuvent être liées à certains troubles du sommeil et à des affections neurologiques.
- Ondes thêta (θ) (4-8 Hz) :
- L’activité thêta est observée pendant la somnolence, la méditation et le sommeil paradoxal (mouvements oculaires rapides).
- L’augmentation des ondes thêta est associée à la pensée créative et à la consolidation de la mémoire.
- Ondes alpha (α) (8-12 Hz) :
- Prédominent pendant l’éveil détendu, les yeux fermés.
- Les ondes alpha sont liées à un état mental calme et alerte.
- Ondes bêta (β) (12-30 Hz) :
- Associées avec l’éveil actif et les tâches cognitives.
- Les fréquences bêta élevées sont associées à une vigilance et une concentration accrues.
- Ondes gamma (γ) (30-100 Hz) :
- Associées aux processus cognitifs de haut niveau, à la perception et à la résolution de problèmes.
- Une activité gamma anormale peut être liée à certains troubles neurologiques.
L’étude de l’interaction de ces bandes EEG fournit des informations précieuses sur le fonctionnement du cerveau, aidant les chercheurs et les cliniciens à comprendre les processus cognitifs, à diagnostiquer les troubles et à mettre au point des interventions thérapeutiques. L’analyse des données EEG permet d’explorer la dynamique cérébrale et peut ouvrir de nouvelles perspectives sur la complexité de l’esprit humain.
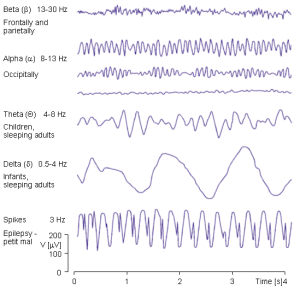
Canaux EEG et positionnement des électrodes
Dans les données EEG (électroencéphalographie), les canaux font référence aux endroits spécifiques du cuir chevelu où des électrodes sont placées pour enregistrer l’activité électrique produite par le cerveau. Ces électrodes font partie d’un capuchon ou d’un réseau EEG. Chaque canal correspond à une électrode unique, et les signaux recueillis à partir de ces canaux fournissent collectivement une vue d’ensemble de l’activité cérébrale.
L’emplacement des canaux est crucial pour la capture des signaux provenant des différentes régions du cerveau. Des normes internationales communes, telles que le système 10-20, sont utilisées pour définir l’emplacement de ces canaux. Ce système doit son nom au fait que la distance entre les électrodes adjacentes représente environ 20 % de la distance totale entre l’avant et l’arrière ou entre la droite et la gauche du crâne, en fonction de la région. Chaque canal enregistre les fluctuations de tension en temps réel, reflétant l’activité électrique des neurones dans la région cérébrale sous-jacente. L’analyse des données EEG provenant de plusieurs canaux permet aux chercheurs et aux cliniciens d’examiner la distribution spatiale de l’activité cérébrale et d’identifier les schémas associés à divers états cognitifs, troubles ou tâches spécifiques.
L’analyse des données EEG avec R
Dans cette section, nous nous concentrons sur l’analyse des données EEG à l’aide de R. Même si l’analyse des données EEG peut être effectuée à la fois avec MATLAB et R, et que le choix entre les deux dépend souvent des préférences du chercheur, de la disponibilité de boîtes à outils ou de packages spécifiques, et de la nature de l’analyse, MATLAB est un choix plus populaire pour l’analyse des données EEG en raison de sa large gamme de packages pour l’analyse des données EEG.
Les outils principaux pour R :
Il existe plusieurs outils pour prétraiter, visualiser et extraire des informations significatives des enregistrements EEG. Vous trouverez ci-dessous un bref aperçu des étapes typiques de l’analyse des données EEG à l’aide de R :
- eegkit: Un outil pour l’importation et le prétraitement des données EEG dans R.
- eegUtils: Un outil pour effectuer le prétraitement de base de l’EEG et le traçage des données EEG.
- ERP: Un logiciel pour analyser, identifier et extraire les potentiels liés à l’événement (ERP) liés à des stimuli ou des événements spécifiques dans R.
Des logiciels clés :
- EEGLAB: boîte à outils MATLAB pour le traitement et l’analyse des données EEG. Elle comprend une variété de fonctions pour l’importation, le prétraitement, la visualisation et l’analyse des données EEG. EEGLAB fournit également une interface utilisateur graphique (GUI) pour l’analyse des données EEG.
- FieldTrip: Boîte à outils MATLAB pour l’analyse des données EEG et MEG. Elle comprend des algorithmes pour l’analyse simple et avancée des données MEG et EEG, tels que l’analyse temps-fréquence, la reconstruction des sources à l’aide de dipôles, de sources distribuées et de formateurs de faisceaux, l’analyse de la connectivité et les tests statistiques non paramétriques.
- Brainstorm: Une boîte à outils MATLAB dédiée à l’analyse des enregistrements cérébraux : MEG, EEG, fNIRS, ECoG, électrodes de profondeur et électrophysiologie multi-unités.
Traitement de l’EEG et analyse statistique en R
Prétraitement de l’EEG
Ce processus vise à éliminer le bruit, les artefacts et autres éléments indésirables tout en préservant l’intégrité des signaux neuronaux. Un prétraitement efficace garantit des résultats fiables lors des analyses ultérieures, en se concentrant sur l’activité neuronale réelle. Voici les principales étapes du prétraitement des données EEG :
- Filtrage :
- L’application de filtres permet d’éliminer les bruits et les artefacts indésirables. Les filtres passe-bas éliminent les bruits à haute fréquence, tandis que les filtres passe-haut suppriment les dérives lentes.
- Des filtres à encoche peuvent être utilisés pour éliminer des fréquences spécifiques, telles que les interférences des lignes électriques.
- Suppression des artefacts :
- Identifier et supprimer les artefacts causés par les mouvements oculaires, l’activité musculaire ou les interférences externes.
- Des techniques telles que l’analyse en composantes indépendantes (ICA) peuvent aider à séparer et à éliminer les artefacts du signal EEG.
- Segmentation :
- Diviser le signal EEG continu en segments plus courts, ce qui facilite l’analyse d’événements ou de tâches spécifiques.
- La segmentation permet aux chercheurs de se concentrer sur des périodes d’intérêt, telles que la présentation d’un stimulus ou les réponses motrices.
- Correction de la ligne de base :
- Ajuster le signal EEG pour obtenir une ligne de base cohérente, souvent en soustrayant le signal moyen sur une période pré-stimulus spécifique.
- La correction de la ligne de base permet de comparer les changements relatifs des amplitudes EEG dans différentes conditions expérimentales.
- Référencement :
- Choisissez une référence appropriée pour les données EEG. Les références les plus courantes sont la référence moyenne ou les mastoïdes liés.
- La référence garantit que les signaux enregistrés reflètent l’activité par rapport à un point défini.
- Interpolation :
- Traiter les canaux manquants ou de mauvaise qualité en interpolant leurs valeurs sur la base des informations relatives aux électrodes environnantes.
- Cette étape permet de maintenir l’intégrité spatiale des données EEG.
- Normalisation :
- Normaliser les amplitudes EEG si nécessaire, afin de faciliter les comparaisons entre différents sujets ou conditions expérimentales.
En mettant en œuvre ces étapes de prétraitement, les chercheurs peuvent améliorer la qualité des données EEG, réduire le bruit et améliorer la précision des analyses ultérieures, ce qui permet d’obtenir des informations plus fiables sur les fonctions cérébrales et la cognition.
Analyse statistique des données EEG
L’analyse statistique des données EEG (électroencéphalographie) est essentielle pour tirer des conclusions significatives des résultats expérimentaux. Les expériences EEG impliquent souvent la comparaison de conditions, de groupes ou de points dans le temps afin de découvrir des modèles d’activité cérébrale associés à des processus cognitifs spécifiques ou à des manipulations expérimentales. Voici un aperçu des principales considérations et méthodes d’analyse statistique des données EEG :
- Statistiques descriptives :
- Utiliser des mesures telles que la moyenne, la médiane et l’écart-type pour fournir un résumé de la tendance centrale et de la variabilité des signaux EEG.
- Les statistiques descriptives permettent une compréhension préliminaire des caractéristiques des données.
- Statistiques inférentielles :
- Appliquez les statistiques inférentielles pour faire des prédictions ou des déductions sur la population dans son ensemble, sur la base des données EEG observées.
- Les tests courants comprennent les tests t, l’ANOVA et l’analyse de régression pour évaluer l’importance des différences entre les conditions ou les groupes.
- Analyse temps-fréquence :
- Utilisez des techniques telles que la transformée de Fourier rapide (FFT) pour analyser le contenu en fréquence des signaux EEG dans le temps.
- L’analyse temps-fréquence permet de comprendre les changements dynamiques de l’activité cérébrale associés à différentes tâches ou stimuli.
- Potentiels liés à l’événement (PFE) :
- Extraire et analyser les ERP pour examiner les réponses neuronales associées à des événements ou des stimuli spécifiques.
- Les méthodes statistiques permettent d’identifier les composantes significatives des ERP et les différences entre les conditions expérimentales.
- Regroupement et classification :
- Utilisez des algorithmes de clustering pour regrouper les modèles EEG et révéler les structures cachées dans les données.
- Les méthodes de classification, telles que les algorithmes d’apprentissage automatique, permettent de distinguer différents états ou conditions cognitifs.
- Analyse des corrélations :
- Explorer les relations entre les caractéristiques de l’EEG et les variables comportementales ou cliniques.
- L’analyse des corrélations permet d’identifier les associations qui contribuent à une compréhension globale des relations entre le cerveau et le comportement.
- Correction des comparaisons multiples :
- Mettre en œuvre des méthodes de correction, telles que Bonferroni ou le taux de fausse découverte (FDR), pour résoudre le problème des taux d’erreur de type I gonflés lors de la réalisation de tests statistiques multiples.
- Cartographie topographique :
- Créez des cartes topographiques pour visualiser les distributions spatiales de l’activité EEG.
- Les analyses statistiques peuvent mettre en évidence des différences significatives entre les régions du cerveau dans diverses conditions expérimentales.
En utilisant ces approches statistiques, les chercheurs peuvent tirer des conclusions solides des données EEG, découvrir des modèles et élucider les mécanismes neurophysiologiques qui sous-tendent les processus cognitifs ou les conditions cliniques.
Utilisation de eegkit pour l’analyse des données EEG en R.
# Install eegkit package
install.packages("eegkit")
# Load eegkit package
library(eegkit)
# Load EEG data
data("eegdata")
# View the first 5 rows of the data
head(eegdata)
eegsmooth Lissage de l’électroencéphalographie (EEG) à un ou plusieurs canaux dans l’espace et/ou le temps.
- Exemple : Lissage des données dans le temp
## get "PZ" electrode of "c" subjects
idx <- which(eegdata$channel=="PZ" & eegdata$group=="c")
eegdata1 <- eegdata[idx,]
## Lissage temporal
eegmod <- eegsmooth(eegdata1$voltage,time=eegdata1$time)
## Définir les données pour la prédiction
time <- seq(min(eegdata1$time),max(eegdata1$time),length.out=100) yhat <- predict(eegmod,newdata=time,se.fit=TRUE) ## visualiser les résultats avec eegtime eegtime(time*1000/255,yhat$fit,voltageSE=yhat$se.fit,ylim=c(-4,4),main="Pz")
 `eegtime` Creates plot of single-channel electroencephalography (EEG) time course with optional confidence
interval. User can control the plot orientation, line types, line colors, etc.
`eegtime` Creates plot of single-channel electroencephalography (EEG) time course with optional confidence
interval. User can control the plot orientation, line types, line colors, etc.
Example: Plotting a single channel
## get "PZ" electrode from "eegdata" data
idx <- which(eegdata$channel=="PZ")
eegdata2 <- eegdata[idx,]
## get average and standard error (note se=sd/sqrt(n))
eegmean <- tapply(eegdata2$voltage,list(eegdata2$time,eegdata2$group),mean)
eegse <- tapply(eegdata2$voltage,list(eegdata2$time,eegdata2$group),sd)/sqrt(50)
## plot results with legend
tseq <- seq(0,1000,length.out=256)
eegtime(tseq,eegmean[,2],voltageSE=eegse[,2],ylim=c(-10,6),main="Pz")
eegtime(tseq,eegmean[,1],vlty=2,vcol="red",voltageSE=eegse[,1],scol="pink",add=TRUE)
legend("bottomright",c("controls","alcoholics"),lty=c(1,2),
lwd=c(2,2),col=c("blue","red"),bty="n")
Références et lectures complémentaires :
- L’atelier en ligne EEGLAB propose un tutoriel sur l’analyse des données EEG dans MATLAB.
- Des informations plus détaillées sur l’analyse des données EEG en R sont disponibles dans la documentation eegkit.
- Pour plus d’informations sur l’analyse des données EEG en Python, consultez la documentation MNE.
- Pour plus d’informations sur l’analyse des données EEG en MATLAB, consultez la documentation EEGLAB
Exemple d’étude EEG statistique : Différences de niveau de tension entre les groupes
Dans une étude portant sur des enregistrements EEG, les données de 10 alcooliques et de 10 sujets témoins ont été recueillies au cours d’une expérience de 10 secondes. L’ensemble de données comprend quatre colonnes : “ID”, “Groupe”, “Temps” et “Tension”.
Analyse exploratoire en R
- Chargez l’ensemble de données EEG à l’aide de la commande read.csv() dans R.
- Dans l’ensemble de données, le groupe est un facteur à deux niveaux : Contrôle et Alcool.
Analyse statistique en R
Effectuez les analyses suivantes à l’aide des données chargées :
- Visualisation de la tension lors du EEG :
- Créez un tracé linéaire pour visualiser la tension EEG au fil du temps pour le participant avec l’ID 1.
- Statistiques descriptives :
- Calculer les statistiques descriptives (moyenne, écart-type, etc.) pour la colonne “Voltage” au sein de chaque groupe (alcooliques et sujets témoins).
- Test T :
- Effectuez un test-t sur des échantillons indépendants pour évaluer s’il existe une différence significative dans les valeurs moyennes de tension entre les alcooliques et les sujets témoins. Tirer des inférences basées sur les résultats.
- Analyse ANOVA :
- Effectuez une analyse de la variance (ANOVA) pour évaluer s’il existe des différences significatives dans les valeurs moyennes de tension entre les groupes. Faites des déductions sur la base des résultats.
Cette étude vise à explorer et à analyser statistiquement les données EEG afin de déterminer s’il existe des différences perceptibles dans les niveaux de tension entre les alcooliques et les sujets témoins. La combinaison de la visualisation exploratoire et des tests statistiques permet une compréhension globale des schémas EEG et des distinctions potentielles entre les groupes.
Fichiers à télécharger :
- eeg.csv
N02: L’imagerie par résonance magnétique (IRM) structurelle
Matin Yousefabadi
L’analyse de l’IRM en R : un guide pédagogique
Introduction à l’imagerie par résonance magnétique (IRM)
L’imagerie par résonance magnétique (IRM) est une technique d’imagerie médicale essentielle qui utilise la résonance magnétique nucléaire pour produire des structures internes détaillées du corps, particulièrement efficaces pour visualiser les tissus mous avec une clarté supérieure à celle des rayons X ou des tomodensitogrammes. Cette modalité est largement utilisée pour diagnostiquer un large éventail d’affections, notamment l’imagerie cérébrale pour détecter les tumeurs, les accidents vasculaires cérébraux et d’autres affections neurologiques.
L’IRM implique que le patient soit allongé à l’intérieur d’un grand aimant, où des ondes radio ciblent le corps. Les capteurs de l’IRM détectent l’énergie émise par le corps et convertissent ces données en images. Contrairement aux méthodes faisant appel aux rayonnements ionisants, le profil de sécurité de l’IRM permet une utilisation répétée. Toutefois, son champ magnétique puissant peut être contre-indiqué chez les patients porteurs d’implants métalliques spécifiques, et certains peuvent trouver la procédure longue et immobile difficile.
Dans le domaine des neurosciences, les capacités d’imagerie non invasives et détaillées de l’IRM sont indispensables pour distinguer avec précision les tissus cérébraux et détecter les anomalies, jouant ainsi un rôle essentiel dans le diagnostic et le suivi de maladies neurologiques telles que la sclérose en plaques et la maladie d’Alzheimer.
Analyse des données d’IRM à l’aide de la programmation R
Dans cette section, nous nous concentrons sur la visualisation et l’analyse des données d’IRM à l’aide de la programmation R.
Format des données :
Les images IRM sont généralement disponibles au format NIFTI, avec des extensions de fichier telles que .nii ou .nii.gz (compressé). Les fichiers NIFTI sont compatibles avec divers logiciels d’analyse en neuroimagerie.
Progiciels R clés :
- oro.nifti : Indispensable pour charger et manipuler les objets NIFTI.
- neurobase : Étend les capacités de oro.nifti, en offrant des fonctions d’imagerie supplémentaires.
Chargement de données IRM dans R :
# Loading the oro.nifti and neurobase packages
library(oro.nifti)
library(neurobase)
# Reading a NIFTI file
mri_img = readnii("training01_01_mri_img.nii.gz")
Visualiser les données de l’IRM
- Les trois plans orthogonaux :
ortho2(mri_img)
cette fonction de neurobase montre un objet nifti en 3 plans.
- Vue en boîte à lumière :
image(mri_img, useRaster= TRUE)
Cette fonction de oro.nifti offre une vue en boîte lumineuse montrant toutes les planches de l’IRM.
- L’observation des planches spécifiques :
oro.nifti::slice(mri_img, z = c(60, 80))
Est nécessaire pour l’examen détaillé de certaines structures neuroanatomiques
Analyse des distributions des valeurs des voxels :
En IRM, les voxels (abréviation de “pixels volumétriques”) fonctionnent de la même manière que les pixels des images 2D, mais ils représentent les plus petites unités tridimensionnelles distinguables du volume scanné.
Les valeurs des voxels dans les données d’IRM peuvent être analysées pour comprendre la distribution des différents types de tissus
- Visualisation de la densité :
plot(density(mri_img))
- ce graphique aide à comprendre la distribution des intensités des voxels
- L’histogramme :
hist(mri_img)
- Les histogrammes sont pratiques pour visualiser la fréquence des différentes valeurs d’intensité des voxels.
La segmentation des régions du cerveau :
Un aspect essentiel de l’analyse IRM en neurosciences c’ est la segmentation du cerveau en régions d’intérêt biologiquement significatives (ROI). Cela comprend la segmentation des tissus, l’identification des structures de la matière grise profonde et la segmentation des pathologies telles que les lésions de sclérose en plaques ou les tumeurs.
Références et lectures complémentaires :
Pour des informations plus détaillées et des techniques avancées d’analyse d’IRM à l’aide de R, les ressources suivantes sont recommandées :
- Les bases de l’interprétation de l’IRM – Cet article fournit une approche systématique de l’interprétation de l’IRM, qui est cruciale pour comprendre les images IRM.
- Cours interactif gratuit sur l’imagerie par résonance magnétiquere – Ce cours en ligne complet est conçu pour expliquer de manière simple le fonctionnement de l’imagerie par résonance magnétique. Il couvre un large éventail de sujets, notamment le spin nucléaire, l’instrumentation et la sécurité de l’IRM, le signal RMN et le contraste de l’IRM, le codage spatial dans l’IRM, la formation de l’image IRM, les séquences, l’amélioration du contraste de l’IRM, la qualité de l’image et les artefacts.
- Analyse de la neuroimagerie avec R – Il s’agit d’un excellent tutoriel sur l’utilisation de R pour l’analyse de l’IRM.
Traitement d’images IRM et analyses statistiques avec R
Prétraiter les images IRM
Le prétraitement est une étape critique de l’analyse des images IRM, essentielle pour garantir la précision et la fiabilité des résultats. Ce processus comprend plusieurs étapes clés :
- Correction des artefacts et réduction du bruit : La correction des artefacts dus aux mouvements du patient et aux erreurs de l’équipement, ainsi que la réduction du bruit, sont essentielles pour obtenir des images claires.
- Normalisation des images : La normalisation des images compense les facteurs physiologiques et les différences dans les protocoles de balayage, ce qui permet une comparaison cohérente entre les différents balayages et les différents sujets.
- Normalisation spatiale et segmentation du cerveau : Ces processus alignent les images sur un espace commun et séparent les tissus cérébraux, respectivement, ce qui est essentiel pour une analyse précise.
- Ajustement des facteurs de confusion : La correction des différents facteurs de confusion garantit que les résultats de l’analyse ne sont pas faussés par des facteurs externes.
Le prétraitement améliore la qualité de l’image, l’interprétabilité et la puissance statistique des analyses. Il prépare le terrain pour des analyses avancées, y compris des études d’IRM fonctionnelle et des approches d’apprentissage automatique.
Bien que le prétraitement soit essentiel, il n’est pas l’objet de ce tutoriel. Pour obtenir des méthodes détaillées de prétraitement des images IRM dans R, nous vous recommandons vivement de consulter ce tutoriel tutorial de John Muschelli et Kristin Linn.
Pour les besoins de ce tutoriel, nous supposerons que le prétraitement est déjà terminé.
L’analyse statistique des images IRM
L’analyse statistique des données d’IRM comprend une variété de techniques, chacune offrant un aperçu unique de la structure et de la fonction du cerveau :
- Analyse basée sur les voxels : Elle consiste à comparer les voxels individuels entre les sujets ou les conditions à l’aide de tests statistiques (par exemple, tests t, ANOVA) afin d’identifier les différences dans la structure ou la fonction du cerveau.
- Analyse des régions d’intérêt (ROI) : Utilise des méthodes statistiques pour comparer les caractéristiques de régions cérébrales prédéfinies, ce qui facilite l’étude de maladies ou de fonctions spécifiques.
- Repérage des formes et apprentissage automatique : Ces méthodes avancées, y compris les algorithmes d’apprentissage automatique, permettent d’identifier des schémas dans les données d’IRM indiquant des maladies ou des conditions spécifiques.
- Analyse longitudinale : Des modèles statistiques sont utilisés pour les études qui suivent l’évolution du cerveau dans le temps, ce qui est essentiel pour comprendre la progression ou le développement d’une maladie.
- Analyse des réseaux : Elle se concentre sur l’analyse de la connectivité cérébrale, en utilisant des méthodes statistiques pour comprendre les réseaux cérébraux complexes et leur perturbation dans les troubles neurologiques.
Morphométrie basée sur les voxels
La morphométrie basée sur les voxels est une technique populaire dans l’analyse IRM. Ce processus consiste à se concentrer sur des régions d’intérêt spécifiques (ROI) dans le cerveau et à analyser le volume de ces régions. Le volume d’une région peut être calculé en multipliant le nombre de voxels dans la région par le volume de chaque voxel.
Volume des régions d’intérêt
Bien que notre objectif principal ne soit pas de calculer le volume d’une zone d’intérêt, les personnes intéressées peuvent suivre les étapes suivantes pour masquer une zone d’intérêt et calculer son volume. Notez que ces étapes nécessitent des logiciels et des bibliothèques spécifiques.
Conditions préalables
Assurez-vous que les progiciels nécessaires sont installés. L’installation d’ANTsR est plus complexe que celle des paquets R habituels. Vous trouverez des instructions d’installation détaillées ici.
Guide Pratique (par étape):
- Chargement des progiciels :
library(ANTsR)
library(oro.nifti)
- Lire l’image IRM (dans le format NifTI) :
mri_image <- niftiImageRead("path_to_your_mri_image.nii", reorient = FALSE)
- Prétraiter l’image :
Ceci comprend la normalisation, la réduction du bruit, et dépend fortement sur vos données et les exigences des analyses en fonction de la question à l’étude.
- Segmentation de l’image :
Ces techniques dépendent de la qualité de vos images, les exigences spécifiques ( ANTsR fournit de nombreux outils pour ces fins). Par exemple, utilisant Atropos ( une méthode de segmentation a plusieurs classes)
segmentation_results <- atropos(a = mri_image, m = '[3,1x1x1]', c = '[2,0]', i = 'kmeans[3]', x = 1)
- Extraire et sauvegarder les images segmentées :
Ceci dépend de comment on définit les étiquettes de segmentation Par exemple
csf <- segmentation_results$segmentation == 1
gm <- segmentation_results$segmentation == 2
wm <- segmentation_results$segmentation == 3
niftiImageWrite(csf, "csf_segmented.nii")
niftiImageWrite(gm, "gm_segmented.nii")
niftiImageWrite(wm, "wm_segmented.nii")
- Calculer le volume de chaque voxel :
vres = voxres(t1, units = "cm")
vol_csf = csf * vres
Un exemple d’une étude statistique: la morphométrie basée sur les voxels et la maladie d’Alzheimer.
Prenons l’exemple d’une étude portant sur 30 adultes de plus de 55 ans atteints de la maladie d’Alzheimer et 30 témoins. L’étude s’étend sur 2 ans et suit les changements de volume de l’hippocampe.
- Chargez le fichier alzheimer_hippo_vol.csv à l’aide de la fonction read.csv() dans R.
- Dans l’ensemble de données, la condition est un facteur à deux niveaux : contrôle et alzheimer.
- Au début de l’étude, les images IRM sont recodées et les volumes de l’hippocampe sont calculés. Le volume est indiqué dans la colonne initial_vol.
- Après 2 ans, les IRM sont refaites et la différence de volume est listée dans la colonne “loss”.
Analyses statistiques en R :
Effectuez les analyses suivantes sur les données que vous avez chargées à votre environnement d’analyse:
1. Représentation graphique des moyennes marginales de diagnostic: visualiser la perte moyenne du volume de l’hippocampe chez les patients atteints de la maladie d’Alzheimer par rapport au groupe de contrôle.
An interactive H5P element has been excluded from this version of the text. You can view it online here:
https://ecampusontario.pressbooks.pub/rspnc/?p=181#h5p-7
2. Analyse de la variance (ANOVA) : Effectuez une ANOVA pour évaluer l’effet de la maladie d’Alzheimer sur la perte de volume. Cette analyse permettra de déterminer si la perte de volume est significativement différente entre les patients atteints de la maladie d’Alzheimer et le groupe de contrôle.
An interactive H5P element has been excluded from this version of the text. You can view it online here:
https://ecampusontario.pressbooks.pub/rspnc/?p=181#h5p-8
3. Defi – l’analyse de la covariance (ANCOVA) : En tant que défi avancé, utilisez l’ANCOVA pour évaluer l’effet de la maladie d’Alzheimer sur la perte de volume tout en contrôlant l’association linéaire entre la perte de volume et le volume initial. Cette analyse tient compte du volume initial de l’hippocampe, ce qui permet de mieux comprendre l’impact de la maladie.
An interactive H5P element has been excluded from this version of the text. You can view it online here:
https://ecampusontario.pressbooks.pub/rspnc/?p=181#h5p-9
Cette étude de cas fournit une application pratique du traitement d’images IRM et de l’analyse statistique en R, démontrant la puissance de ces techniques dans la compréhension de conditions neurologiques complexes telles que la maladie d’Alzheimer.
Fichiers à télécharger :
- alzheimer_hippo_vol.csv
N03 : L’IRM Fonctionnelle
Matin Yousefabadi
Introduction à l’imagerie par résonance magnétique fonctionnelle (IRMf)
L’imagerie par résonance magnétique fonctionnelle (IRMf) est une technique de neuro-imagerie non invasive qui a révolutionné notre compréhension du cerveau. Elle est principalement utilisée pour observer et mesurer l’activité cérébrale et constitue un outil précieux pour la recherche en neurosciences. Elle est principalement utilisée pour observer et mesurer l’activité cérébrale, ce qui en fait un outil précieux pour la recherche en neurosciences. L’IRMf fonctionne selon le principe que le flux sanguin cérébral et l’activation neuronale sont couplés. Lorsqu’une zone du cerveau est plus active, elle consomme plus d’oxygène et, pour répondre à cette demande accrue, le flux sanguin vers la zone active augmente également. Ce phénomène est connu sous le nom de réponse hémodynamique.
Les notions de base : L’IRMf
1. Le principe d’opération :
- Contraste dépendant du niveau d’oxygénation du sang (BOLD) : l’IRMf utilise principalement le contraste BOLD, qui repose sur les différentes propriétés magnétiques du sang oxygéné et désoxygéné. Le sang oxygéné (qui est moins magnétique) et le sang désoxygéné (qui est plus magnétique) affectent différemment le signal RM, ce qui permet de détecter les changements du flux sanguin liés à l’activité neuronale.
2. La procédure d’imagerie :
- Non invasive et sécuritaire : l’IRMf utilise un champ magnétique puissant et des ondes radio pour générer des images détaillées du cerveau. Contrairement à d’autres techniques d’imagerie, elle n’implique pas d’exposition au rayonnement ionisant, ce qui la rend sécuritaire pour un usage répété.
- Résolution temporelle et spatiale : Tout en offrant une résolution spatiale relativement élevée (capacité à détecter l’endroit où l’activité se produit dans le cerveau), l’IRMf a une résolution temporelle modeste (capacité à détecter le moment où l’activité se produit). Cela est dû au temps nécessaire pour que la réponse hémodynamique se produise après l’activité neuronale.
3. Les données d’IRMf :
- Les données d’IRMf : Les données brutes de l’IRMf se présentent généralement sous la forme d’images ou de volumes en 3D. Ceux-ci sont acquis au fil du temps, ce qui donne un ensemble de données 4D (espace 3D + temps).
Les applications de l’IRM en recherches neuroscientifiques
1. La cartographie neurologique :
- Identification des zones fonctionnelles : l’IRMf est largement utilisée pour cartographier les zones fonctionnelles du cerveau. Il s’agit notamment de localiser les régions responsables des fonctions motrices, du langage, de la vision et d’autres processus cognitifs.
2. Comprendre les syndromes et les maladies neurologiques :
- Diagnostic et traitement des maladies : Les chercheurs utilisent l’IRMf pour étudier les fonctions cérébrales des personnes souffrant de divers troubles neurologiques et psychiatriques, ce qui facilite le diagnostic et les stratégies de traitement.
3. Les études cognitives et comportementales :
- Aperçu des processus cognitifs : l’IRMf permet aux scientifiques d’observer le cerveau pendant qu’il traite l’information, ce qui donne un aperçu des processus cognitifs complexes tels que la mémoire, l’attention et la résolution de problèmes.
4. La neuroplasticité:
- Suivi des changements au fil du temps : l’IRMf peut être utilisée pour étudier les changements de l’activité cérébrale au fil du temps, ce qui permet de comprendre la neuroplasticité, c’est-à-dire la capacité du cerveau à se réorganiser en formant de nouvelles connexions neuronales.
Des limitations et des défis:
1. Les mesures indirectes :
- La réponse BOLD est une mesure indirecte de l’activité neuronale, qui s’appuie sur les variations du flux sanguin plutôt que sur la mesure directe des potentiels d’action neuronale.
2. La résolution temporelle :
- la réponse hémodynamique et naturellement lente, et ceci limite la précision temporelle de l’IRMf
3. Les artefacts et le bruit :
- Les données de l’IRMf peuvent être affectées par différents types de bruits et d’artefacts, y compris les mouvements du patient et les processus physiologiques tels que la respiration et le rythme cardiaque.
4. Le coût et l’accessibilité :
- L’IRMf est une technique coûteuse qui nécessite un équipement et une expertise spécialisés, ce qui limite son accessibilité.
Des outils communement utilises pour l’analyse des données d’IRMf :
- SPM (Statistical Parametric Mapping) :
- Principalement basé sur MATLAB, SPM est un outil largement utilisé pour analyser les données d’imagerie cérébrale. Il se concentre sur l’analyse statistique des fonctions cérébrales à l’aide de méthodes basées sur le voxel.
- FSL (FMRIB Software Library) :
- FSL est un ensemble de progiciels complets d’outils d’analyse pour les données d’imagerie cérébrale FMRI, MRI et DTI. Elle est connue pour ses pipelines de prétraitement robustes et ses capacités d’analyse statistique avancée.
- AFNI (Analysis of Functional NeuroImages) :
- AFNI est une suite de programmes en C pour le traitement, l’analyse et l’affichage de données d’IRM fonctionnelle (IRMf). Il est particulièrement performant dans l’analyse des séries temporelles pour examiner les changements dans l’activité cérébrale.
- FreeSurfer :
- FreeSurfer est principalement utilisé pour le traitement et l’analyse de données de neuroimagerie structurelle et fonctionnelle provenant d’IRM. Il excelle dans la segmentation du cerveau et la reconstruction de la surface corticale.
- nilearn (Python) :
- Nilearn est un module Python pour l’apprentissage statistique rapide et facile des données de neuro-imagerie. Il s’appuie sur scikit-learn et convient aux approches d’apprentissage automatique en neuro-imagerie.
Le prétraitement des données d’IRMf :
Le prétraitement des données IRMf est essentiel pour améliorer la qualité des données, normaliser les données d’une session à l’autre et d’un sujet à l’autre, supprimer les artefacts dus aux mouvements du sujet et aux processus physiologiques, et optimiser l’interprétation du signal BOLD, garantissant ainsi la précision et la fiabilité des analyses ultérieures.
Les étapes du prétraitement des données d’IRMf:
- Correction de la synchronisation des planches :
- Corriger la différence de temps dans l’acquisition de l’image entre les différentes planches imagées du cerveau. Cette étape est importante car toutes les coupes ne sont pas acquises simultanément.
- Correction du mouvement :
- Corriger les mouvements de la tête du sujet pendant l’examen. Même de petits mouvements peuvent affecter de manière significative la qualité des données.
- Normalisation spatiale :
- Transformer toutes les images cérébrales en un espace commun (souvent un modèle cérébral standard comme le modèle MNI), ce qui permet d’effectuer des comparaisons entre les sujets.
- Lissage :
- Applique un filtre spatial aux données afin d’augmenter le rapport signal/bruit. Le lissage rend les données moins bruyantes mais peut aussi brouiller les détails les plus fins.
- Filtrage temporel :
- Supprimer les fluctuations qui ne sont pas liées à la réponse hémodynamique du cerveau, telles que le bruit à haute fréquence ou les dérives à basse fréquence du signal.
- Détection et correction des artefacts :
- Identifier et corriger les artefacts physiologiques tels que les battements cardiaques et la respiration, ainsi que d’autres événements sporadiques susceptibles de fausser les données.
- Co-registrement :
- Aligner les images fonctionnelles avec les images structurelles (comme les scans pondérés en T1) pour assurer une localisation précise de l’activité cérébrale.
L’analyse de l’IRMf avec R
Dans cette section, nous nous concentrerons sur la manière dont vous pouvez visualiser et travailler avec des données d’IRMf dans R.
Bien que les données d’IRMf puissent être enregistrées dans des formats bruts tels que DICOM ou PAR/REC spécifique au scanner, les types les plus courants pour l’analyse sont les formats traités tels que le NIfTI, largement utilisé, qui stocke les données et informations sur le volume cérébral, et BIDS, une structure de répertoire standardisée facilitant le partage des données et la compatibilité avec divers outils d’analyse. Le choix du bon type dépend de l’étape de l’analyse et des besoins de compatibilité, mais NIfTI et BIDS sont généralement préférés pour les données traitées en raison de leur flexibilité et de leur adoption généralisée. Pour ce tutoriel, nous utilisons les formats de fichiers NIFTI.
Vous pouvez télécharger ici un exemple de données IRMf enregistrées au format NIFTI.
1. Chargement des progiciels necessaires :
install.packages(c("oro.nifti", "fmri", "neurobase", "fslr"))
library(oro.nifti)
library(fmri)
library(neurobase)
library(fslr)
2. Chargement des donnees d’IRMf :
# Load a NIfTI file
fmri_data <- readNIfTI("path/to/your/fmri.nii")
# Check the dimensions and structure
dim(fmri_data) # Check dimensions (x, y, z, time)
# As you can see fMRI has 4 dimensions which are 3D space + time.
str(fmri_data) # View data structure
3. Visualisation des donnees d’IRMf :
- Visualisation a partir des planches :
# Display a single slice
ortho2(fmri_data, xyz = c(40, 40, 20)) # Visualize slice at coordinates (40, 40, 20)
- Visualisation en serie temporelle :
- En IRMf, un voxel est un minuscule morceau de cerveau en 3D, comme un pixel dans une image. Il mesure les variations du flux sanguin liées à l’activité cérébrale, ce qui nous donne une image détaillée de ce qui se passe où et quand.
# Extract time series from a specific voxel.
voxel_time_series <- fmri_data[25, 30, 15, ]
# Plot the time series
plot(voxel_time_series, type = "l", xlab = "Time", ylab = "BOLD Signal")
Un exemple d’une étude : les réponses neuronales aux stimuli visuels
Dans une étude de neuro-imagerie, une expérience d’IRMf a été menée pour étudier les réponses du cerveau à des stimuli visuels. L’étude a utilisé un paradigme en blocs dans lequel les participants ont vu des images d’un bébé à des intervalles fixes. Plus précisément, chaque participant a été exposé à l’image d’un bébé pendant 15 secondes, suivies d’un intervalle de 15 secondes pendant lequel aucune image n’était affichée (écran noir). Cette séquence a été répétée pendant 10 cycles, soit une durée totale de l’expérience de 300 secondes, ou 5 minutes.
L’ensemble de données voxels.csv qui l’accompagne contient des séries temporelles de données provenant de 10 voxels cérébraux sélectionnés d’un participant, fournissant un aperçu ciblé de l’activité cérébrale localisée au cours de l’expérience. Les données sont structurées de manière à faciliter l’analyse des schémas de réponse du cerveau en relation avec le stimulus visuel. En outre, l’ensemble de données comprend une colonne de stimuli qui indique le moment où les stimuli visuels ont été présentés au participant. Dans cette colonne, une valeur de 1 indique la présence de l’image du bébé à l’écran, tandis qu’une valeur de 0 indique une phase où aucune image n’a été montrée (écran noir). Les données de l’IRMf ont été enregistrées toutes les secondes, il y a donc 300 pas de temps dans les données.
Certainement ! Voici quelques questions que vous pourriez poser aux étudiants en rapport avec chaque partie du code R fourni, afin de tester leur compréhension de l’analyse et de la visualisation des données en R :
Chargement des données
- Écrivez le code R pour charger un fichier CSV nommé “voxels.csv” dans une variable appelée voxels_data. Expliquez ce que fait chaque partie de la commande.
Inspection des données
- Comment afficher les premières lignes du jeu de données voxels_data dans R ? Pourquoi cette étape est-elle importante avant de procéder à l’analyse des données ?
- Quelle fonction utiliseriez-vous pour comprendre la structure de voxels_data ? Quel type d’informations cette fonction fournit-elle ?
Preparation des données
- Dans le cadre de l’analyse des données, pourquoi est-il important de préparer ou de nettoyer vos données avant de procéder à l’analyse statistique ? Donnez un exemple d’étape de préparation des données que vous pourriez avoir à effectuer pour cet ensemble de données.
Analyse statistique
- Écrivez un extrait de code pour calculer la corrélation entre la série temporelle de chaque voxel et les stimuli. Expliquez comment la fonction apply est utilisée dans ce contexte.
- Que fait la fonction cor() ? Dans ce contexte spécifique, qu’est-ce que nous essayons de découvrir en utilisant cor() ?
Visualisation et représentations graphiques de données
- Créez un diagramme à barres en R à l’aide de ggplot2 qui affiche les coefficients de corrélation pour chaque voxel. Expliquez comment vous avez défini l’esthétique des x et des y dans votre graphique.
- Pourquoi la visualisation est-elle importante dans l’analyse des données, en particulier dans le contexte de cette étude de neuro-imagerie ?
Fichiers à télécharger :
- voxels.csv
Références et lectures complémentaires :
Pour des informations plus détaillées et des techniques avancées d’analyse statistique de l’IRMf à l’aide de R, les ressources suivantes sont recommandées :
- Validation des méthodes d’IRMf
- Tutoriel sur la modélisation des données d’IRMf à l’aide d’un modèle linéaire général. – Un exemple très complet d’analyse statistique de l’IRMf à l’aide de R
N04: Le traitement des données
Maheshwar Panday
Vous êtes-vous déjà demandé comment interpréter un ensemble de données ? Parfois, les ensembles de données sont disponibles mais dans des formats qui semblent déroutants ? Parfois, le format d’organisation dans lequel vous recevez un ensemble de données n’a pas beaucoup de sens et n’est pas très utile pour vous renseigner sur les données. Pour obtenir des informations sur vos données, vous devez parfois utiliser des outils d’organisation des données – en R, nous appelons cette série d’outils “data wrangling”.
En chargeant la série de paquets tidyverse – un groupe de paquets (tidyr, dplyr et ggplot2), nous pouvons facilement organiser, ordonner et visualiser nos données afin de vérifier qu’elles sont organisées dans des formats raisonnables.
Dans cet article, nous allons passer en revue quelques-unes des opérations de base de tidyverse qui sont parmi les plus utilisées, et les utiliser pour manipuler un ensemble de données catégoriques afin de présenter l’information de manière succincte.
Quelques ressources pratiques : voici quelques feuilles de contrôle pour les notions de base pour le nettoyage et le traitement des données dans RStudio en utilisant la série de paquets tidyverse (N.B. ces feuilles de contrôle sont développés par posit) :
Data Wrangling : https://www.rstudio.com/wp-content/uploads/2015/02/data-wrangling-cheatsheet.pdf
Tidyr : https://bioinformatics.ccr.cancer.gov/docs/rintro/resources/tidyr_cheatsheet.pdf
Dplyr : https://nyu-cdsc.github.io/learningr/assets/data-transformation.pdf
Chargement des progiciels
library(tidyverse)
library(dplyr)
library(ggplot2)
library(RColorBrewer)
library(here)
library(gridExtra)
Les données utilisées pour cette série d’exercices tidyverse proviennent du dépôt de données d’apprentissage automatique de l’Université de Californie à Irvine (UC Irvine Machine learning). Des informations sommaires sur l’ensemble de données et les variables qu’il contient sont disponibles sur le lien suivant : https://archive.ics.uci.edu/dataset/915/differentiated+thyroid+cancer+recurrence
D’autres informations sur l’ensemble de données et son utilisation sont disponibles dans le document source
Borzooei, S., Briganti, G., Golparian, M. et al. Machine learning for risk stratification of thyroid cancer patients: a 15-year cohort study. Eur Arch Otorhinolaryngol (2023). https://doi.org/10.1007/s00405-023-08299-w
Comprendre l’opérateur pipe %>% et comment l’utiliser pour pouvoir manier vos données
La première chose à comprendre est l’opérateur pipe %>%. Ce petit champion de la manipulation de données vous permet d’écrire proprement et d’enchaîner une série d’opérations de manipulation de données afin d’effectuer de manière transparente une série de manipulations de données pour produire la structure souhaitée de l’image de données avec les colonnes et les lignes nécessaires pour produire les visualisations qui présentent le mieux les informations contenues dans l’image de données.
Tout d’abord, l’opérateur pipe peut être appliqué aux vecteurs ainsi qu’aux cadres de données. De la même manière que nous pouvons “canaliser” les sorties de vecteurs dans des opérations, nous pouvons “canaliser” des colonnes ou des cadres de données entiers dans des fonctions de manipulation de données afin de produire la structure de données requise.
Pour commencer la prochaine série de questions, vous devrez être à l’aise avec l’opérateur pipe pour manipuler vos données à partir du cadre de données sur le cancer de la thyroïde que vous avez chargé dans RStudio. La première activité consiste à manipuler le cadre de données pour compter le nombre de cas de cancer de la thyroïde pour chacune des quatre pathologies différentes de l’ensemble de données.
no.piped.mean <- mean(c(1,2,3,4,5,6,7,8,9, 10, 11, 12))
piped.mean <- c(1,2,3,4,5,6,7,8,9,10,11, 12) %>% mean()
print (paste("mean without pipe operator: ", no.piped.mean,
"mean with pipe operator: ", piped.mean))
thyroid.by.pathology <- thyroid.data %>% group_by(Pathology)
print (head(thyroid.by.pathology))
pathology.frequencies<- thyroid.by.pathology %>% summarise (Frequency = n())
print (head(pathology.frequencies))
L’organisation visuelle des données, obtenir des renseignements sur vos données à partir de vos données
Cet ensemble de données contient 4 pathologies cancéreuses uniques : Folliculaire, Cellule de Hurthel, Micropapillaire et Papillaire. Combien de cas de chaque type se trouvent dans cet ensemble de données ? Présentez vos résultats sous la forme d’un histogramme de fréquence. Annotez les barres de l’histogramme de fréquence pour indiquer le nombre de cas dans chaque pathologie.
Conseil : utilisez la série de packages tidyverse pour manipuler vos données.
pathology_freq <- thyroid.data %>%
group_by(Pathology) %>%
summarise(Frequency = n())
print(pathology_freq)
thyroid.cancer.freqplot <- ggplot(pathology_freq, aes(x = Pathology, y = Frequency)) +
geom_bar(stat = "identity", fill = "lightsteelblue3") +
geom_text(aes(label = Frequency), vjust = 1.5, hjust = 0.5) +
theme(axis.text.x = element_text(angle = 45, hjust = 1)) +
theme_classic()+
labs(x = "Pathology", y = "Frequency", title = "Frequency Distribution : Pathology Categories")
print (thyroid.cancer.freqplot)
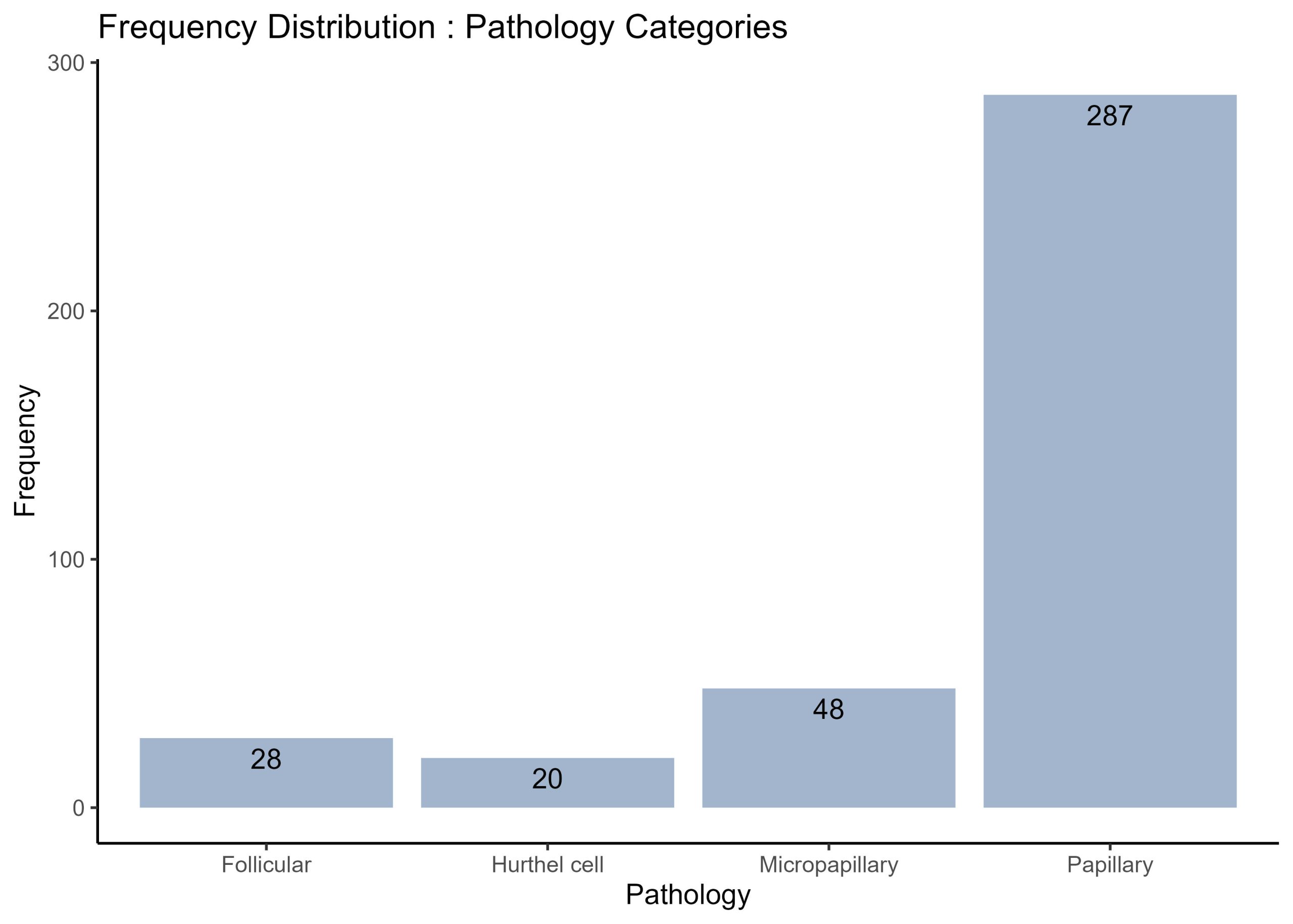
Le premier histogramme de fréquence est intéressant car il permet de visualiser facilement le nombre de cas de chaque type de pathologie cancéreuse dans l’ensemble de données, mais il est important de noter qu’il existe d’autres données sur les patients qui sont également précieuses, mais qui ne sont pas présentes dans la visualisation précédente. Supposons que vous souhaitiez également connaître le nombre d’hommes et de femmes pour chaque pathologie. Comment obtiendrez-vous les proportions d’hommes et de femmes pour chaque pathologie et comment modifieriez-vous l’histogramme de fréquence ci-dessus pour visualiser le nombre d’hommes et de femmes pour chaque groupe de pathologie ?
Modifiez l’histogramme de fréquence ci-dessus pour montrer les proportions d’hommes et de femmes dans chaque pathologie, et annotez chaque barre avec les nombres d’hommes et de femmes dans chaque groupe de pathologie.
gender_pathology_freq <- thyroid.data %>%
group_by(Pathology, Gender) %>%
summarise(Frequency = n())
thyroid.freqplot.by.Gender <- ggplot(gender_pathology_freq, aes(x = Pathology, y = Frequency, fill = Gender)) +
geom_bar(stat = "identity") +
geom_text(aes(label = Frequency), position = position_stack(vjust = 0.5)) +
theme(axis.text.x = element_text(angle = 45, hjust = 1)) +
theme_classic()+
labs(x = "Pathology", y = "Frequency", fill = "Gender", title = "Frequency Distribution of Each Pathology Category by Gender")
print (thyroid.freqplot.by.Gender)
Visualiser les différentes proportions d’examens physiques pour catégorie de pathologie
Les histogrammes de fréquence sont un moyen utile de visualiser les nombres et les proportions, mais supposons que vous souhaitiez voir la proportion d’un ensemble de conditions diagnostiques dans une série de pathologies cancéreuses. Pour chaque patient, l’examen physique de la glande thyroïde a été classé dans l’une des cinq catégories suivantes : goitre diffus, goitre multinodulaire, normal, goitre nodulaire unique gauche et goitre nodulaire unique droit.
Il existe quatre pathologies distinctes (comme vous l’avez appris dans l’exercice précédent). Supposons que vous exploitiez ces données et que vous souhaitez connaître les proportions de chaque catégorie de diagnostic physique pour chacune des pathologies. Oui, vous pouvez utiliser un histogramme de fréquence comme développé précédemment, mais vous pouvez également utiliser un diagramme circulaire pour rendre les proportions des catégories de diagnostic physique facilement visibles.
Préparez une série de 4 diagrammes circulaires – un diagramme pour chaque pathologie et, dans chaque diagramme, indiquez la proportion de cas pour chaque catégorie d’examen physique.
colour.palette <- c("maroon3", "mediumslateblue", "olivedrab3", "cadetblue2", "darkgoldenrod2" )
pathology_exam_freq <- thyroid.data %>%
group_by(Pathology, Physical.Examination) %>%
summarise(Frequency = n())
total_pathology <- pathology_exam_freq %>%
group_by(Pathology) %>%
summarise(Total = sum(Frequency))
pathology_exam_freq <- left_join(pathology_exam_freq, total_pathology, by = "Pathology")
pathology_exam_freq <- pathology_exam_freq %>%
mutate(Proportion = Frequency / Total)
Diagnosis.by.Pathology <- pathology_exam_freq %>%
ggplot(aes(x = "", y = Proportion, fill = Physical.Examination)) +
geom_bar(width = 1, stat = "identity") +
geom_text(aes(label = Frequency), position = position_stack(vjust = 0.5)) +
coord_polar("y", start = 0) +
facet_wrap(~Pathology) +
theme_void() +
theme(legend.position = "right") +
scale_fill_manual(values = colour.palette)+
labs(fill = "Physical Examination", title = "Proportion of Cases in Each Physical Examination Category by Pathology")
print (Diagnosis.by.Pathology)
Il s’agit ci-dessus d’un bon moyen de montrer combien de caractéristiques d’examen physique sont présentes dans chaque pathologie cancéreuse. Maintenant, comme vous l’avez fait pour l’histogramme de fréquence ci-dessus, séparez ces données pour montrer les proportions des catégories d’examen physique dans chaque pathologie cancéreuse, par sexe. Cette fois, vous devez créer deux séries de 4 graphiques. Une série pour les hommes et une autre pour les femmes. Chaque graphique montre la proportion de catégories d’examens physiques pour une pathologie donnée.
colour.palette <- c("maroon3", "mediumslateblue", "olivedrab3", "cadetblue2", "darkgoldenrod2" )
gender_pathology_exam_freq <- thyroid.data %>%
group_by(Gender, Pathology, Physical.Examination) %>%
summarise(Frequency = n())
total_gender_pathology <- gender_pathology_exam_freq %>%
group_by(Gender, Pathology) %>%
summarise(Total = sum(Frequency))
gender_pathology_exam_freq <- left_join(gender_pathology_exam_freq, total_gender_pathology, by = c("Gender", "Pathology"))
gender_pathology_exam_freq <- gender_pathology_exam_freq %>%
mutate(Proportion = Frequency / Total)
Diagnosis.by.Pathology.by.Gender<- gender_pathology_exam_freq %>%
ggplot(aes(x = "", y = Proportion, fill = Physical.Examination)) +
geom_bar(width = 1, stat = "identity") +
geom_text(aes(label = Frequency), position = position_stack(vjust = 0.5)) +
coord_polar("y", start = 0) +
facet_grid(Gender ~ Pathology) +
theme_void() +
theme(legend.position = "top",
plot.title = element_text(hjust = 0.5)) +
scale_fill_manual(values = colour.palette)+
labs(fill = "Physical Examination", title = "Proportion of Cases for Each Physical Examination Category by Pathology and Gender")
print (Diagnosis.by.Pathology.by.Gender)
Donnez des diagrammes en boîte de l’âge par sexe pour chaque diagnostic physique par pathologie
La grammaire de manipulation des données fournie par les progiciels de base de tidyverse constitue un moyen essentiel de regrouper et d’organiser vos données afin de visualiser les tendances, les effectifs et les proportions plus simplement, au moyen de visualisations convaincantes et succinctes.
Un excellent exemple est la création d’une série de diagrammes en boîte. La série de 8 diagrammes circulaires que vous venez de générer dans le dernier exercice montre les proportions des catégories d’examen physique pour chaque pathologie et pour chaque sexe. Mais tous les patients de cet ensemble de données n’ont pas le même âge. Il serait important de connaître également les données démographiques relatives à l’âge. Créez une série de diagrammes en boîte montrant l’âge par sexe pour chaque catégorie d’examen physique pour chaque pathologie cancéreuse. Il s’agit d’une série de 20 diagrammes en boîte.
Cette fois, nous devons utiliser des diagrammes en boîte plutôt que des diagrammes circulaires ou des histogrammes de fréquence, puisque nous voulons montrer l’éventail des âges par sexe pour chaque groupe de catégories.
thyroid.age.boxplots <- thyroid.data %>%
ggplot(aes(x = Gender, y = Age, fill = Gender)) +
geom_boxplot() +
facet_grid(Pathology ~ Physical.Examination) +
theme(axis.text.x = element_text(angle = 45, hjust = 1)) +
labs(x = "Gender", y = "Age", fill = "Gender", title = "Boxplots of Age by Gender for Each Physical Examination Category by Pathology")
print (thyroid.age.boxplots)
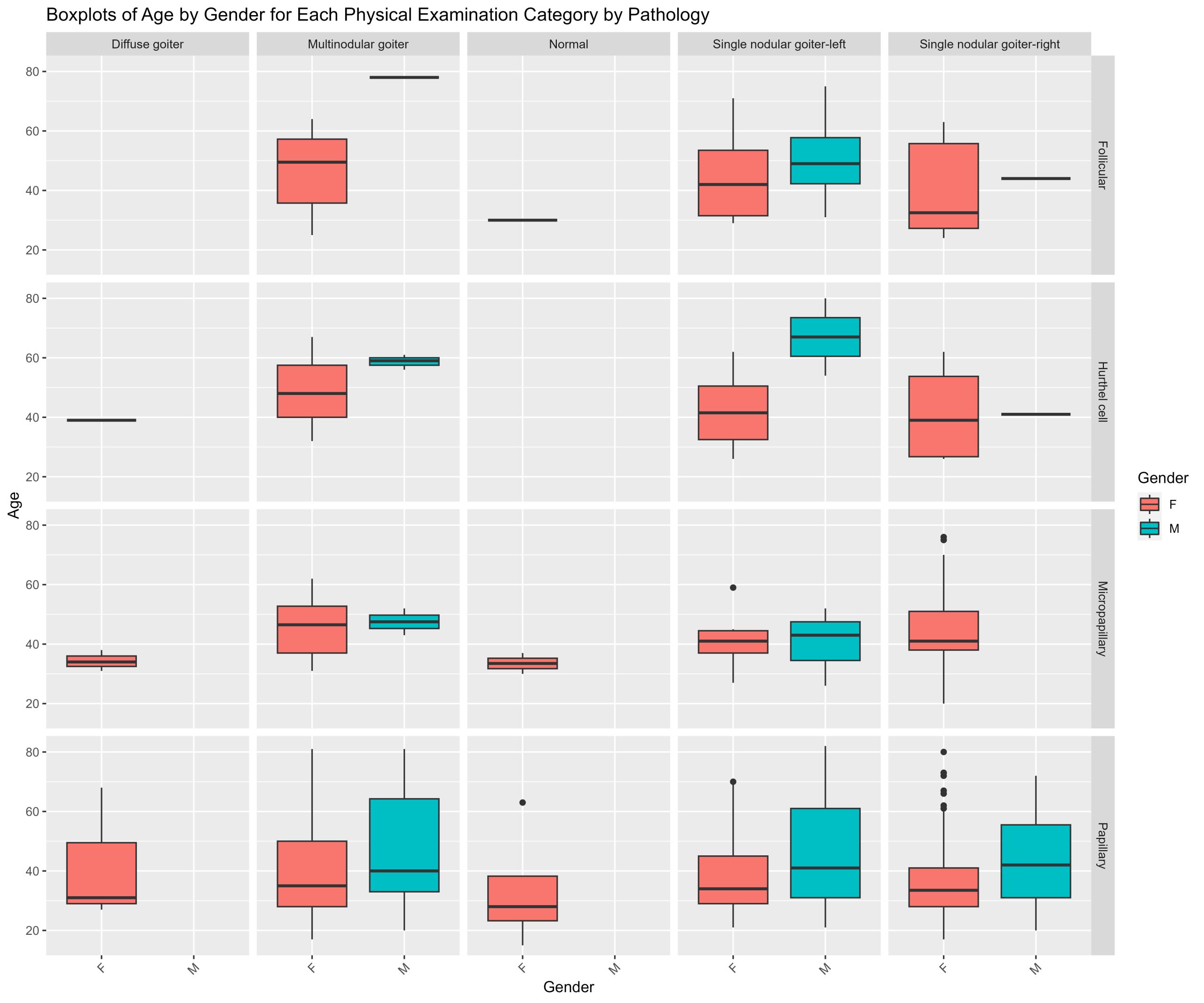
Des diagrammes circulaires pour chaque caractéristique
Bien que vous puissiez préparer une série d’effectifs, de proportions ou de diagrammes en boîte pour diverses combinaisons de caractéristiques imbriquées les unes dans les autres. Une étape essentielle consiste à comprendre quelles sont vos caractéristiques, ce que signifient les informations catégorielles contenues dans chaque caractéristique et quelles proportions de vos données se retrouvent dans chaque caractéristique. Vous pouvez y parvenir en créant des diagrammes circulaires pour chaque caractéristique, à l’exception de l’âge du patient. Vous devriez probablement effectuer cette opération au début avant d’entreprendre votre propre analyse d’un ensemble de données catégorielles, mais ce processus est un peu plus compliqué et nous le réservons donc pour la fin. Le processus implique à la fois une série d’opérations de manipulation et des outils de visualisation.
C’est à partir d’analyses exploratoires telles que les graphiques générés dans cette grille de diagrammes circulaires que le chaînage des informations sur les caractéristiques de la pathologie, du sexe, de l’examen physique et de l’âge a semblé intéressant !
data_without_age <- thyroid.data[ , !(names(thyroid.data) %in% "Age")]
plot_list <- list()
for (column_name in names(data_without_age)) {
proportions <- data_without_age %>%
group_by(.data[[column_name]]) %>%
summarise(n = n()) %>%
mutate(prop = n / sum(n))
pie_chart <- ggplot(proportions, aes(x = "", y = prop, fill = .data[[column_name]])) +
geom_bar(width = 1, stat = "identity") +
coord_polar("y", start = 0) +
labs(title = paste("Pie Chart for", column_name), x = NULL, y = NULL, fill = column_name) +
theme(plot.margin = margin (1,1,1,1, "cm"))+
theme_void()
plot_list[[column_name]] <- pie_chart
}
plotgrid <- grid.arrange(grobs = plot_list, ncol = 4, returnGrob= TRUE)
Fichiers à télécharger :
Pour télécharger, cliquez avec le bouton droit de la souris et appuyez sur “Enregistrer le fichier sous” ou “Télécharger le fichier lié”.
- Thyroid_Diff.csv
N05: Les données à haute-dimension
Maheshwar Panday
Données sur le cancer du sein au Wisconsin : Analyse des données à haute dimension
Vous êtes pathologiste et vous avez mesuré 569 noyaux de cellules à partir de prélèvements à l’aiguille de masses de tissu mammaire. Les échantillons proviennent de masses bénignes (B) ou malignes (M). Vous souhaitez effectuer une analyse de la forme et de la taille des cellules entre les cellules bénignes et malignes afin de mieux comprendre les différences qui existent entre elles. Vous aimeriez également explorer l’utilisation de l’apprentissage automatique pour voir dans quelle mesure de simples algorithmes de regroupement peuvent identifier les cellules bénignes des cellules malignes en se basant UNIQUEMENT sur leurs caractéristiques de taille et de forme. Utilisation de l’ensemble de données sur le cancer du sein du Wisconsin
—- Texte tiré de Kaggle ——-
Les données sur le cancer du sein comprennent 569 exemples de biopsies cancéreuses, chacune comportant 32 caractéristiques. Une caractéristique est un numéro d’identification, une autre est le diagnostic du cancer et 30 sont des mesures de laboratoire à valeur numérique. Le diagnostic est codé “M” pour malin ou “B” pour bénin.
Les 30 autres mesures numériques comprennent la moyenne, l’erreur standard et la valeur la plus mauvaise (c’est-à-dire la plus grande) pour 10 caractéristiques différentes des noyaux cellulaires numérisés, qui sont les suivantes:-
Rayon, Texture ,Périmètre, Surface, Lisse Compacité, Concavité, Concave Points, Symétrie ,Dimension fractale. Les données sont disponisbles du : https://archive.ics.uci.edu/dataset/17/breast+cancer+wisconsin+diagnostic
Pour en savoir plus sur l’ensemble de données du Wisconsin sur le cancer du sein, et plus particulièrement sur la manière dont chacune des caractéristiques de cet ensemble de données a été calculée, veuillez consulter :
Street, W.N., Wolberg, W.H., & Mangasarian, O.L. (1993). Nuclear feature extraction for breast tumor diagnosis. Electronic imaging.
(1) chargement de progiciels de statistiques, de regroupement, de réduction de la dimensionnalité et de visualisation des données
Installez et chargez les progiciels suivants dans votre environnement de RStudio.
library(dabestr)
library(ggplot2)
library(pheatmap)
library(tidyverse)
library(dplyr)
library(stats)
library(RColorBrewer)
library(Rtsne)
(2) Chargement des données
Téléchargez les données à partir du UCI Machine Learning Repository. Charger ensuite le fichier csv des mesures des cellules dans votre environnement de RStudio.
BreastCancer.Data <- read.csv("WisconsinBreastCancerData.csv")
BreastCancer.Data$X <- NULL
print (head(BreastCancer.Data))
(3) préparer les cadres de données pour le tsne et le regroupement ultérieur
L’ensemble de la base de données BreastCancer.Data est structuré de telle sorte que chaque ligne correspond à une cellule et chaque colonne à un paramètre. Toutefois, certaines colonnes ne sont pas des caractéristiques (descriptions quantitatives des cellules). Lorsqu’une colonne n’est pas une caractéristique, il s’agit d’une étiquette. Les étiquettes sont des moyens d’identifier ou de marquer des cellules spécifiques une fois que nous avons compris comment elles sont caractérisées par leurs caractéristiques.
Avant de pouvoir explorer l’ensemble de données, nous devons séparer les étiquettes des caractéristiques. Deux colonnes du cadre de données étiquettent les cellules – id -> le numéro d’identification unique attribué à une cellule – diagnostic -> si la cellule provient d’un échantillon bénin (B) ou malin (M)).
Les colonnes restantes sont des caractéristiques qui servent de descriptions quantitatives de la taille et de la forme des cellules.
- Votre première tâche avec ces données est de subdiviser l’ensemble de la base de données sur le cancer du sein en deux bases de données
- Diagnostic.Labels – qui contiendra les métadonnées
- Diagnostic.Features – qui contiendra les caractéristiques
- Étant donné que les amplitudes des mesures s’étendent sur des échelles différentes, vous devez placer vos données dans un espace commun pour permettre aux variations et aux différences de devenir apparentes entre les caractéristiques de l’ensemble de l’ensemble des données. Obtenez la cote-Z votre ensemble de données à l’aide de l’opération scale() dans R sur votre Diagnostic.Features.
Diagnostic.Labels <- select(BreastCancer.Data, id, diagnosis)
feature.columns <- setdiff (colnames(BreastCancer.Data), colnames(Diagnostic.Labels))
Diagnostic.Features <- BreastCancer.Data[, feature.columns]
Diagnostic.Features.Scored <- scale(Diagnostic.Features)
view(Diagnostic.Features.Scored)
print (colnames(Diagnostic.Features.Scored)
(4) explorer l’ensemble de données à l’aide de t-SNE
Tsne ou le T-distributed Stochastic Neighbour Embedding est une technique utilisée pour visualiser des données de haute dimension en 2 dimensions. Qualifiée de méthode de réduction de la dimensionnalité, elle nous permet d’explorer les données une caractéristique à la fois. Dans cet ensemble de données sur le cancer du sein, 32 mesures décrivent la taille, la forme et la texture des masses dans le tissu mammaire, qui sont ensuite considérées comme bénignes (B) ou malignes (M).
Pour plus d’informations sur T-sne, veuillez consulter : Van der Maaten, L., Hinton, G. Visualizing Data using T-sne. Journal of Machine Learning Research 9 (2008) 2579-2605
Utilisons tsne pour visualiser la répartition de ces caractéristiques dans l’ensemble des données.
N.B. J’ai exécuté cette boucle for de combinaisons de semences par perplexité car il est important de tester ces paramètres lorsque l’on travaille avec T-sne. La perplexité contrôle la part des éléments de la structure de données locale par rapport aux éléments de la structure de données globale qui contribuent à la visualisation finale lorsque les données passent par la réduction de la dimensionnalité. Les variations de perplexité pour la même graine aléatoire peuvent avoir des effets très importants sur la visualisation finale, même si les données ne changent pas.
perplexities <- c(10, 15, 20, 25, 30)
seeds <- c(123, 456, 789, 246, 135 )
tsne_data <- data.frame()
for (i in 1:length(perplexities)) {
for (j in 1:length(seeds)) {
tsne_model <- Rtsne(Diagnostic.Features.Scored, perplexity = perplexities[i], seed = seeds[j])
tsne_temp <- data.frame(
tSNE1 = tsne_model$Y[, 1],
tSNE2 = tsne_model$Y[, 2],
Perplexity = perplexities[i],
Seed = seeds[j],
Diagnosis = Diagnostic.Labels$diagnosis
)
tsne_data <- rbind(tsne_data, tsne_temp)
}
}
tsne.stampcollection <- ggplot(tsne_data, aes(x = tSNE1, y = tSNE2, colour = Diagnosis)) +
geom_point() +
facet_wrap(Perplexity ~ Seed, scales = "free") +
theme_bw()
(4) Exercice de l’étudiant – tsne pour explorer les cellules bénignes vs malignes en utilisant la réduction de dimensionnalité
Utilisez les paramètres suivants dans votre tsne initial : – seed <- 789 – perplexité <- 30
Construisez une carte T-sne de la base de données Diagnostic.Features. Tracez la carte T-sne résultante à l’aide de ggplot en veillant à colorer les points en fonction de leur étiquette de diagnostic. Que remarquez-vous sur la carte T-sne lorsqu’elle est annotée par diagnostic ?
En guise d’exercice, variez la perplexité pour la graine de 789, en essayant d’aller de 10 à 50 par sauts de 10. Que remarquez-vous sur la carte T-sne lorsque vous faites varier la perplexité ? Selon vous, que contrôle le paramètre de perplexité dans l’algorithme T-sne ?
set.seed (789)
perplexity <- 30
seed <- 789
tsne_model <- Rtsne(Diagnostic.Features.Scored, perplexity = perplexity, seed = seed)
tsne_data <- data.frame(
tSNE1 = tsne_model$Y[, 1],
tSNE2 = tsne_model$Y[, 2],
Diagnosis = Diagnostic.Labels$diagnosis
)
library(ggplot2)
tsne.by.diagnosis <- ggplot(tsne_data, aes(x = tSNE1, y = tSNE2, colour = Diagnosis)) +
geom_point() +
labs( x = "T-SNE Component 1", y= "T-SNE Component 2")
theme_bw()
print(tsne.by.diagnosis)
5) Regroupement hiérarchique d’échantillons malins
Et s’il existait un moyen d’identifier facilement les cellules comme étant bénignes ou malignes en fonction de leurs paramètres de taille et de forme ? Les algorithmes de regroupement sont un exemple d’approche fondée sur les données pour regrouper vos données sur la base des modèles présents dans les caractéristiques. Bien sûr, les données contiennent des étiquettes, mais que se passerait-il si vous faisiez des regroupements sur les valeurs des caractéristiques, les mesures, sans fournir d’informations sur le diagnostic des cellules ? Sélectionnez 2 grappes – nous voulons voir si le regroupement hiérarchique permet de distinguer les cellules bénignes des cellules malignes en se basant uniquement sur les informations relatives à leur forme et à leur taille.
Les étapes du regroupement hiérarchique sont les suivantes :
- Construire une matrice de dissimilarité des caractéristiques a partir des cotes- z. Utilisez les distances euclidiennes
- Appelez la fonction hclust sur la matrice de dissimilarité et spécifiez que la méthode est ward.D2
- Annotez le dendrogramme pour montrer où les données sont réparties en groupes (clusters)
- Visualisez les résultats de votre regroupement en construisant une carte thermique de la matrice de dissimilarité organisée avec un dendrogramme entouré et annoté.
num.clusters <- 2
dissimilarity.matrix <- dist(Diagnostic.Features.Scored, method = "euclidean")
h.clusters <- hclust(dissimilarity.matrix, method = "ward.D2")
clusters <- cutree(h.clusters, k = num.clusters)
All.Samples.zscored <- cbind(Diagnostic.Labels,
Diagnostic.Features.Scored)
All.Samples.zscored$hclust_clusters <- clusters
annotation.df <- data.frame(Cluster = as.factor(clusters))
rownames(annotation.df) <- rownames(Diagnostic.Features)
k.cluster.colourpalette <- c("olivedrab2", "maroon3")
colourmapping <- setNames(k.cluster.colourpalette, levels (annotation.df$Cluster))
annotation.colours = list(Cluster = k.cluster.colourpalette)
names(annotation.colours$Cluster) <- levels(annotation.df$Cluster)
div.spectral.red.blue <- c("#4a100e", "#731331", "#a52747", "#c65154", "#e47961", "#f0a882", "#fad4ac", "#ffffff",
"#bce2cf", "#89c0c4", "#5793b9", "#397aa8", "#1c5796", "#163771", "#10194d" )
div.spectral.blue.red <- rev(div.spectral.red.blue)
continuous.spectral.redblue <- colorRampPalette(div.spectral.red.blue) (256)
continuous.spectral.bluered <- colorRampPalette(div.spectral.blue.red)(256)
palette.breaks <- seq(from= 0, to = 8, length.out = length (continuous.spectral.redblue) + 1)
hclust.dissimilarity.heatmap <- pheatmap(dissimilarity.matrix,
cluster_rows = h.clusters,
cluster_cols = h.clusters,
annotation_row = annotation.df,
annotation_colors = annotation.colours,
color = continuous.spectral.redblue,
breaks = palette.breaks)
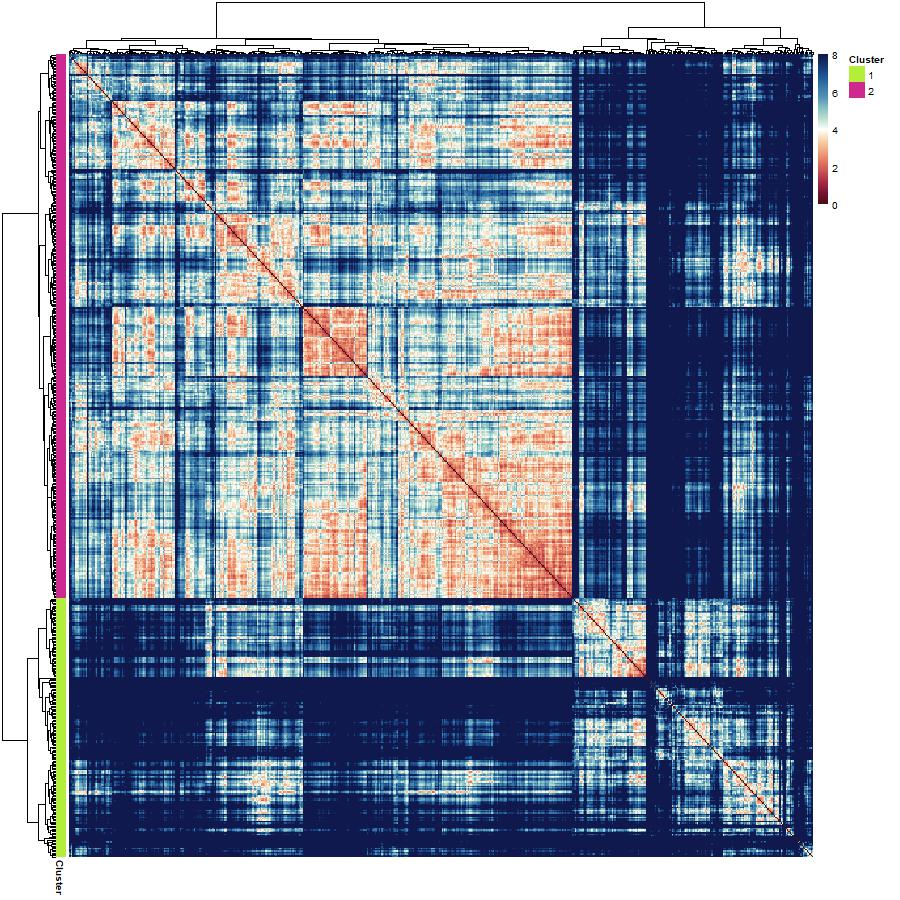
(6) Inspecter les résultats de votre regroupement hiérarchique :
Vous venez de produire un résultat de regroupement hiérarchique. Vous pouvez visualiser le regroupement à l’aide d’une carte thermique et d’un dendrogramme annoté, mais cela ne vous permet pas de savoir quelles cellules ont été reléguées dans quel regroupement. Vous pouvez également produire un diagramme à barres de proportions empilées qui vous indique la proportion de cellules bénignes et malignes dans un groupe.
Préparez un cadre de données de proportions – regroupez les données par groupe et par diagnostic et calculez les pourcentages.
préparez un diagramme à barres de proportions empilées à l’aide de ggplot et de l’élément geom_bar. D’après les résultats du regroupement, dans quelle mesure le regroupement hiérarchique a-t-il permis de distinguer les cellules bénignes des cellules malignes ?
– Question de l’étudiant – Parmi les caractéristiques de votre cadre de données, il existe des modèles qui permettent de distinguer les cellules bénignes des cellules malignes. Vous souhaitez évaluer la différence entre les cellules bénignes et les cellules malignes.
library(dplyr)
All.Samples.zscored.grouped <- All.Samples.zscored %>%
group_by(hclust_clusters, diagnosis) %>%
summarise(n = n()) %>%
mutate(freq = n / sum(n))
view (All.Samples.zscored.grouped)
Hclust.barplot <- ggplot(All.Samples.zscored.grouped, aes(fill=diagnosis, y=freq, x=hclust_clusters)) +
geom_bar(position="fill", stat="identity") +
theme_minimal() +
labs(x="Cluster", y="Proportion", fill="Diagnosis") +
scale_x_continuous(breaks = c(1,2))+
theme(axis.text.x = element_text(angle = 90, hjust = 1))
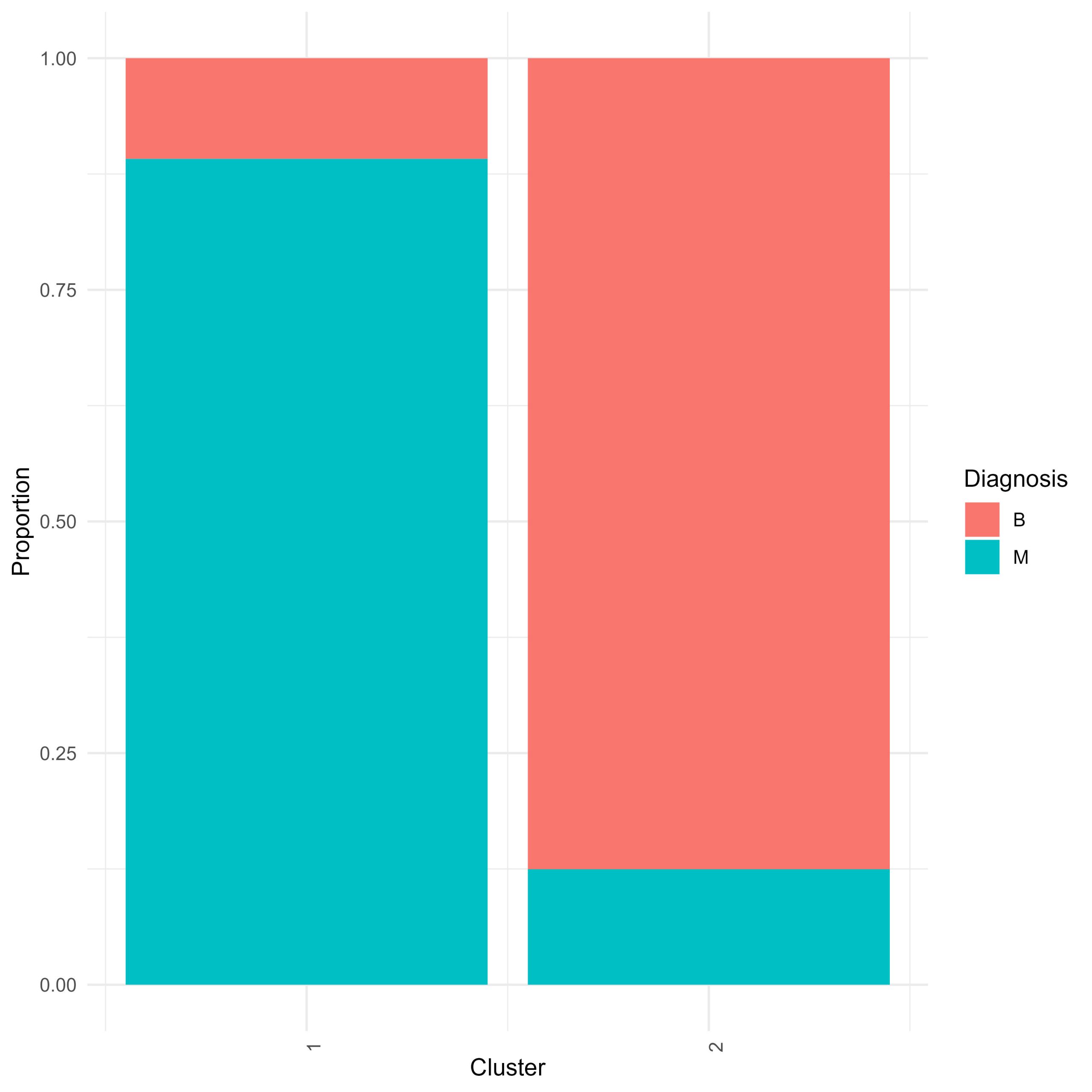
(9) Comparer le regroupement hiérarchique aux étiquettes de diagnostic avec la réduction de la dimensionnalité
Une autre façon d’évaluer l’efficacité de l’affectation des grappes en fonction de l’étiquette de diagnostic consiste à annoter le graphique T-SNE que vous avez construit précédemment, mais cette fois-ci, au lieu de colorer les points en fonction de leur étiquette de diagnostic connue, vous pouvez les annoter en fonction de leurs grappes définies de façon hiérarchique.
Utilisez la même perplexité et la même graine aléatoire que précédemment. De cette façon, vous pouvez comparer la façon dont les grappes de grappes sont représentées dans l’espace à haute dimension et la façon dont les étiquettes de diagnostic sont représentées dans le même espace. Lorsque vous comparez les graphiques t-SNE annotés par diagnostic et par grappe hiérarchique, que remarquez-vous ? Quelles sont les similitudes et les différences qui vous semblent évidentes ?
set.seed (789)
perplexity <- 30
seed <- 789
tsne_model <- Rtsne(Diagnostic.Features.Scored, perplexity = perplexity, seed = seed)
tsne_data <- data.frame(
tSNE1 = tsne_model$Y[, 1],
tSNE2 = tsne_model$Y[, 2],
hclust_cluster = as.factor(All.Samples.zscored$hclust_clusters)
)
view( tsne_data)
library(ggplot2)
hclust.colours <- c("maroon3", "olivedrab2")
tsne.by.hcluster <- ggplot(tsne_data, aes(x = tSNE1, y = tSNE2, colour = hclust_cluster)) +
geom_point() +
labs( x = "T-SNE Component 1", y= "T-SNE Component 2") +
theme_bw() +
scale_color_manual(values = hclust.colours)
print(tsne.by.hcluster)
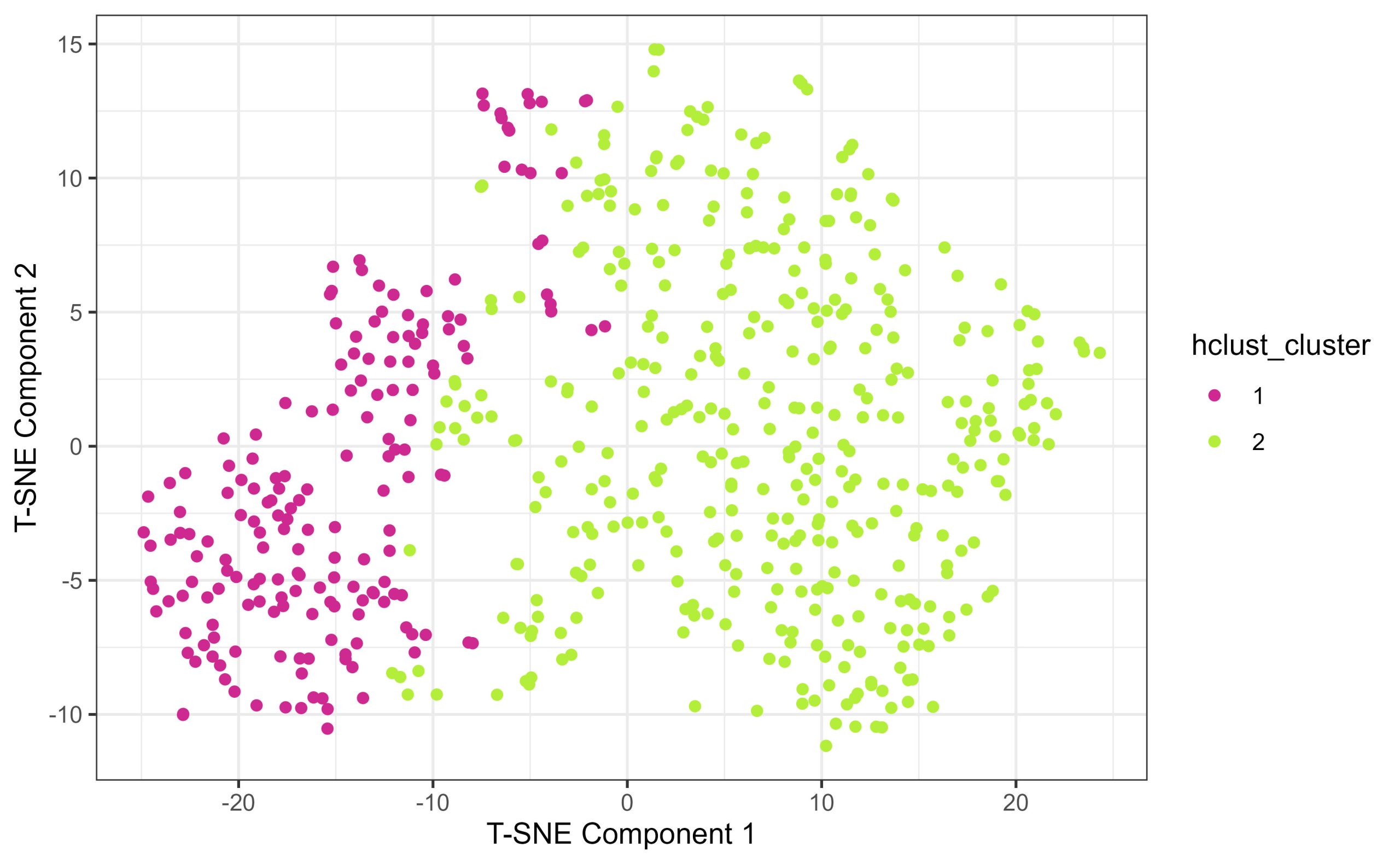
(10) Quelques derniers éléments de réflexion
(réflexion des élèves – peut donner lieu à une discussion sur les algorithmes de regroupement et sur les raisons pour lesquelles il est important de vérifier et de valider les résultats des regroupements)
Vous avez comparé les véritables étiquettes diagnostiques des cellules cancéreuses du sein – bénignes et malignes – à des clusters hiérarchiques construits uniquement à partir de caractéristiques de taille et de forme. Compte tenu de l’efficacité avec laquelle l’algorithme de regroupement a séparé les cellules bénignes et malignes, réfléchissez à l’utilité de cette approche si vous ne saviez pas a priori que les cellules étaient classées dans les catégories de diagnostic bénigne et maligne. Après les avoir regroupées en deux groupes et avoir comparé leurs statistiques, que devriez-vous faire pour confirmer que les cellules d’un groupe sont malignes et que les autres sont bénignes ?
Fichiers à télécharger :
- WisconsinBreastCancerData.csv
Recherche en Comportement
III
C01 : La forme physique des punaises de lit femelles
Brendan McEwen
La forme physique des punaises de lit femelles
Vous êtes un biologiste évolutionniste étudiant le conflit sexuel et l’effet de la fréquence des accouplements sur la condition physique des femelles tout au long de leur vie. Votre système d’étude est le tristement célèbre punaise de lit Climex lectularis. Les punaises de lit vivent en agrégations mixtes où les femelles sont sujettes à un harcèlement reproductif fréquent de la part des mâles. En raison de ce harcèlement reproductif, les femelles varient dans leurs taux d’accouplement dans les groupes naturels où certaines femelles s’accouplent relativement rarement, et d’autres beaucoup plus fréquemment. Des taux d’accouplement plus élevés pour les femelles augmentent le nombre de descendants produits par unité de temps, mais peuvent imposer une pénalité de longévité car le processus physique de reproduction est coûteux. Ce compromis pose la question de savoir si la variation du taux d’accouplement entraîne un changement de la condition physique globale tout au long de la vie (nombre de descendants viables produits). Pour répondre à cette question, vous avez soumis les femelles à un traitement de fréquence d’accouplement élevée (High) ou faible (Low). Vous avez compté le nombre total de descendants viables que chaque femelle a produit au cours de sa vie (Hatchlings), ainsi que sa durée de vie totale en jours (Longevity). Vous avez enregistré vos résultats dans le dataframe contenu dans “femalefitness.csv”.
- Charger les données. Créez une nouvelle colonne appelée FemaleID, qui contient une étiquette d’identification unique pour chaque ligne. Réorganisez les colonnes de manière à ce que FemaleID soit la colonne la plus à gauche de l’ensemble de données.
An interactive H5P element has been excluded from this version of the text. You can view it online here:
https://ecampusontario.pressbooks.pub/rspnc/?p=203#h5p-64
- Produisez deux graphiques à boîtes :
- Une en utilisant les graphiques de base de R, montrant la variation du nombre de larves produites par les femelles qui s’accouplent à basse fréquence par rapport à celles qui s’accouplent à haute fréquence
- Une en utilisant ggplot, montrant la variation de la longévité des femelles qui s’accouplent à basse fréquence par rapport à celles qui s’accouplent à haute fréquence. Distinguez entre les traitements de sorte que la boîte des femelles à basse fréquence soit de couleur vert clair, et celle des femelles à haute fréquence soit de couleur bleu clair.
An interactive H5P element has been excluded from this version of the text. You can view it online here:
https://ecampusontario.pressbooks.pub/rspnc/?p=203#h5p-65
- Créez des histogrammes séparés des larves produites pour les femelles traitées à basse et haute fréquence d’accouplement. Ajustez les limites de l’axe des x de sorte que les histogrammes soient imprimés à la même échelle.
An interactive H5P element has been excluded from this version of the text. You can view it online here:
https://ecampusontario.pressbooks.pub/rspnc/?p=203#h5p-66
- En utilisant le test statistique le plus simple possible qui convient à ce scénario, vérifiez si le taux d’accouplement a un effet sur la condition physique des femelles tout au long de leur vie. Expliquez verbalement l’hypothèse nulle et donnez une déclaration inférentielle pour votre résultat.
An interactive H5P element has been excluded from this version of the text. You can view it online here:
https://ecampusontario.pressbooks.pub/rspnc/?p=203#h5p-67
- En utilisant le test statistique le plus simple possible qui convient à ce scénario, vérifiez si le taux d’accouplement a un effet sur la longévité des femelles.
An interactive H5P element has been excluded from this version of the text. You can view it online here:
https://ecampusontario.pressbooks.pub/rspnc/?p=203#h5p-68
- Calculez le rapport de cotes entre une femelle dans le traitement à faible fréquence d’accouplement vivant plus de 70 jours et une femelle dans le traitement à haute fréquence d’accouplement vivant plus de 70 jours. Donnez une explication verbale de ce que cela signifie réellement. An interactive H5P element has been excluded from this version of the text. You can view it online here:
https://ecampusontario.pressbooks.pub/rspnc/?p=203#h5p-69
Fichiers à télécharger :
Pour télécharger, faites un clic droit et appuyez sur “Enregistrer le fichier sous” ou “Télécharger le fichier lié”
- femalefitness.csv
Laboratoire et Institution ou Investigateur Principal :
Cognitive Ecology Lab, Dr. Reuven Dukas, Département de Psychologie, Neuroscience, & Comportement de l’Université McMaster https://psych.mcmaster.ca/dukas/index.htm
Références et lectures complémentaires :
Yan, J. L., & Dukas, R. (2022). The social consequences of sexual conflict in bed bugs: social networks and sexual attraction. Animal Behaviour, 192, 109-117.
Parker, G. A. (2006). Sexual conflict over mating and fertilization: an overview. Philosophical Transactions of the Royal Society B: Biological Sciences, 361(1466), 235-259.
Maklakov, A. A., Bilde, T., & Lubin, Y. (2005). Sexual conflict in the wild: elevated mating rate reduces female lifetime reproductive success. the american naturalist, 165(S5), S38-S45.
C02 : L'agression des grenouilles
Brendan McEwen
L’agression des grenouilles
Vous êtes un écologiste comportemental intéressé par la territorialité et l’agression chez les grenouilles. La grenouille venimeuse à cuisses brillantes Allobates femoralis dépend de la présence de petites mares d’eau pour sa reproduction. Les mâles de cette espèce se livrent à une compétition territoriale pour sécuriser les zones qui contiennent ces mares d’eau. On ignore cependant à quel moment du développement des grenouilles le comportement territorial émerge. Pour tester cela, vous avez voyagé jusqu’à un site de terrain en Équateur oriental et capturé 50 A. femoralis de divers stades de développement. Vous avez enregistré le sexe de chaque grenouille, mesuré la longueur du museau au cloaque (SVL ; en mm) de chaque grenouille, puis soumis les grenouilles à un test de miroir. Dans le test de miroir, vous avez enregistré si elles exprimaient des comportements agressifs (par exemple, des charges, des morsures ou des luttes) envers leur reflet dans le miroir.
Note : Ce scénario implique une analyse statistique plus avancée connue sous le nom de “Régression logistique”, non couverte ni par les statistiques descriptives ni par les statistiques inférentielles. Le principe général de la régression logistique est de déterminer si une variable prédictive a un effet sur le résultat d’une variable de réponse catégorielle. Dans ce scénario, notre résultat catégoriel est binaire (pas d’agression, 0, versus agression, 1). Cela fait de ce scénario plus spécifiquement une “Régression binomiale”, qui est un sous-ensemble de la régression logistique. Pour une introduction sur la façon de réaliser une régression binomiale en R, voir :
https://bookdown.org/ndphillips/YaRrr/logistic-regression-with-glmfamily-binomial.html
- Chargez les données. Imprimez la plage de SVL pour les mâles et la plage de SVL pour les femelles.
An interactive H5P element has been excluded from this version of the text. You can view it online here:
https://ecampusontario.pressbooks.pub/rspnc/?p=205#h5p-70
- En utilisant la commande glm() en R, déterminez s’il y a une association entre le sexe ou la taille du corps et le comportement agressif chez ces grenouilles. Ne pas ajuster une interaction entre la taille du corps et le sexe.
An interactive H5P element has been excluded from this version of the text. You can view it online here:
https://ecampusontario.pressbooks.pub/rspnc/?p=205#h5p-71
- Pour le sexe et la taille du corps, rédigez une déclaration inférentielle basée sur les résultats de votre modèle de régression binomiale qui inclut à la fois le sexe et la taille du corps.
- Créez un modèle de régression binomiale en utilisant uniquement SVL comme variable prédictive, et enregistrez-le dans un objet appelé “mod”.
An interactive H5P element has been excluded from this version of the text. You can view it online here:
https://ecampusontario.pressbooks.pub/rspnc/?p=205#h5p-72
- En utilisant les graphiques de base de R, tracez les valeurs prédites pour un vecteur de longueurs de museau-cloaque théoriques de 5mm à 30mm, en utilisant le modèle de régression binomiale SVL.
An interactive H5P element has been excluded from this version of the text. You can view it online here:
https://ecampusontario.pressbooks.pub/rspnc/?p=205#h5p-73
Fichiers à télécharger :
Pour télécharger, faites un clic droit et appuyez sur “Enregistrer le fichier sous” ou “Télécharger le fichier lié”
- FrogAggro.csv
Laboratoire et Institution ou Investigateur Principal :
Laboratoire d’Écologie Comportementale et Sensorielle, Dr. James B. Barnett, Département de Zoologie, Trinity College Dublin, Irlande
Références et lectures complémentaires :
Chaloupka, S., Peignier, M., Stückler, S., Araya-Ajoy, Y., Walsh, P., Ringler, M., & Ringler, E. (2022). Repeatable territorial aggression in a Neotropical poison frog. Frontiers in ecology and evolution, 10, 881387.
Rodríguez, C., Fusani, L., Raboisson, G., Hödl, W., Ringler, E., & Canoine, V. (2022). Androgen responsiveness to simulated territorial intrusions in Allobates femoralis males: evidence supporting the challenge hypothesis in a territorial frog. General and comparative endocrinology, 326, 114046.
C03: La coloration des grenouilles
Brendan McEwen
La coloration des grenouilles
Vous êtes un écologiste visuel étudiant la coloration d’avertissement et le mimétisme chez les grenouilles à fléchettes empoisonnées. Votre système d’étude est l’Ameerega bilinguis toxique et l’Allobates zaparo non toxique, une paire d’espèces de grenouilles terrestres sympatriques originaires de l’Amazonie équatorienne. L’Ameerega bilinguis utilise un signal d’avertissement à plusieurs composants, avec un dos rouge, des taches lumineuses jaunes sur les membres et un ventre bleu vif. L’Allobates zaparo a évolué pour imiter cette coloration, mais présente un “mimétisme imparfait” – les propriétés quantitatives exactes des composants de couleur ne sont pas parfaitement assorties. Vous avez collecté 20 individus adultes de chaque espèce sur le terrain et pris des photographies calibrées en couleur de chacune de leurs régions corporelles. Vous avez ensuite utilisé la micaToolbox dans ImageJ pour simuler la vision aviaire et calculer la force du contraste chromatique (c’est-à-dire la teinte) et achromatique (c’est-à-dire la luminosité) de chacun des quatre composants de couleur (Front Spots, Back Spots, Dorsum, Venter) contre un arrière-plan naturel de feuilles mortes, pour chaque espèce. Les valeurs de contraste visuel sont présentées dans l’unité de ‘JND’, ou ‘Just Noticeable Difference’, où des valeurs plus élevées indiquent que la tache de couleur contraste plus fortement avec l’arrière-plan. En d’autres termes, des valeurs plus élevées signifient que le composant du signal est plus visible.
- Chargez les données. Créez des histogrammes pour le contraste chromatique et achromatique de chaque composant (Front Spot, Back Spot, Dorsum, Venter) avec les données des deux espèces disposées sur le même panneau (2 figures au total ; 4 panneaux par figure, 16 histogrammes au total).
- Utilisez la couche facet_wrap() dans ggplot2 pour créer un panneau séparé pour chaque région de couleur dans la même figure.
- Faites remplir le modèle Am. bilinguis en turquoise, et le mimétique Al. zaparo en rouge..
- Rendez les remplissages des deux espèces semi-transparents afin que tout chevauchement potentiel de distribution soit apparent.
- Étiquetez l’axe des x “Contraste de couleur (JND)” et “Contraste de luminance (JND)” respectivement.
An interactive H5P element has been excluded from this version of the text. You can view it online here:
https://ecampusontario.pressbooks.pub/rspnc/?p=207#h5p-74
- En utilisant la fonction ddply() (progiciel : “plyr”) pour créer un cadre de données récapitulatif pour chacune des valeurs de contraste de couleur et de luminance, avec des colonnes d’espèces, de composants, et de n, ainsi que des valeurs moyennes de JND, sd, se, Ci.lwr, et Ci.upr pour le contraste chromatique et achromatique.
An interactive H5P element has been excluded from this version of the text. You can view it online here:
https://ecampusontario.pressbooks.pub/rspnc/?p=207#h5p-75
- En utilisant le dataframe récapitulatif, créez une figure visualisant les contrastes chromatiques (axe des x) et achromatiques (axe des y) moyens des composants du signal de chaque espèce
- Utilisez le package ggplot pour créer un nuage de points
- Coloriez les points par espèce, avec le modèle en cyan et le mimétique en rouge
- Séparez les régions de couleur par la forme du point
- Créez une ligne verticale en pointillés et une ligne horizontale en pointillés, chacune avec une intercept = 3
- Faites en sorte que l’échelle des x et des y soit identique, pour montrer la relation entre les valeurs de contraste achromatique et chromatique
- Enregistrez le graphique sous le nom “AvianBackgroundContrasts”
An interactive H5P element has been excluded from this version of the text. You can view it online here:
https://ecampusontario.pressbooks.pub/rspnc/?p=207#h5p-76
- Effectuez une paire dANOVAs factorielles (1 pour chaque contraste chromatique et achromatique) pour déterminer si le contraste de fond est affecté par le composant du signal, l’espèce, ou une interaction entre les deux.
- En utilisant les figures d’histogrammes comme guides, calculez des tests de LSD de suivi entre le modèle et le mimétique pour les régions qui semblent différer dans leur contraste de fond par espèce. Signalez les différences directionnelles, c’est-à-dire le mimétisme imparfait directionnel par zaparo.
An interactive H5P element has been excluded from this version of the text. You can view it online here:
https://ecampusontario.pressbooks.pub/rspnc/?p=207#h5p-77
Fichiers à télécharger :
Pour télécharger, faites un clic droit et appuyez sur “Enregistrer le fichier sous” ou “Télécharger le fichier lié”
- FrogJNDs.csv
Laboratoire et Institution ou Investigateur Principal :
Laboratoire d’Écologie Comportementale et Sensorielle, Dr. James B. Barnett, Département de Zoologie, Trinity College Dublin, Irlande
https://www.jbbarnett.co.uk/
Références et lectures complémentaires :
McEwen, B.L., Yeager, J.D., Kinley, I., Anderson, H.M., Barnett, J.B.B. (Under Review). Body posture and viewing angle modulate detectability and mimic fidelity in a poison frog system.
Darst, C. R., & Cummings, M. E. (2006). Predator learning favours mimicry of a less-toxic model in poison frogs. Nature, 440(7081), 208-211.
Stevens M, Párraga CA, Cuthill IC, Partridge JC, Troscianko TS. (2007). Using digital photography to study animal coloration. Biol. J. Linn. 90, 211-237
C04: Les lézards envahissants
Brendan McEwen
Les lézards envahissants
Vous êtes un herpétologiste intéressé par les invasions biologiques dans les habitats urbains. L’anole brun Anolis sagrei est une espèce invasive en Floride du Sud, où il est maintenant en concurrence avec l’anole vert indigène Anolis carolinensis. Les mâles des deux espèces se battent pour le territoire dans leur habitat désormais partagé. Les mâles signalent agressivement en utilisant une série de pompes pour montrer leur condition physique aux autres mâles. Les mâles qui font plus de pompes sont considérés comme plus agressifs que les mâles qui en font moins. Vous vous demandez si les anoles bruns envahissants sont de meilleurs compétiteurs spatiaux que les anoles verts indigènes, et si la compétition spatiale chez ces espèces est liée à leur comportement agressif. Pour tester cela, vous avez transporté des individus capturés à l’état sauvage des deux espèces au laboratoire. Vous avez administré trois tours d’un essai d’agression, dans lequel le lézard a été présenté à un miroir et enregistré. Les mâles perçoivent leur reflet comme un intrus et se livrent à un comportement de démonstration de pompes. Vous avez enregistré le nombre de pompes effectuées lors de chacun des trois essais séparés, puis calculé un niveau d’agression moyenne pour chaque lézard. Vous avez ensuite placé un A. carolinensis indigène et un A. sagrei envahissant dans une arène et enregistré la zone d’espace qu’ils occupaient au cours de trois jours (cm3) dans une variable appelée “utilisation de l’espace”, comme mesure de leur capacité à rivaliser pour l’espace.
Analysez cet ensemble de données pour voir si l’ID de l’espèce ou le niveau d’agression a un effet sur la capacité de compétition spatiale.
- Exécutez le bloc de code suivant, sans modification, pour simuler cet ensemble de données.
An interactive H5P element has been excluded from this version of the text. You can view it online here:
https://ecampusontario.pressbooks.pub/rspnc/?p=209#h5p-78
- En utilisant la commande lm(), créez et analysez un modèle statistique qui teste l’effet de l’espèce, de l’agression, et de leur interaction sur l’utilisation de l’espace transformée en racine carrée.
- Donnez une déclaration inférentielle verbale pour chaque effet dans ce modèle.
An interactive H5P element has been excluded from this version of the text. You can view it online here:
https://ecampusontario.pressbooks.pub/rspnc/?p=209#h5p-79
- En utilisant la commande lm(), créez et analysez un modèle statistique qui teste l’effet de l’espèce, de l’agression, et de leur interaction sur l’utilisation de l’espace transformée en racine carrée. Donnez une déclaration inférentielle verbale pour chaque effet dans ce modèle.
An interactive H5P element has been excluded from this version of the text. You can view it online here:
https://ecampusontario.pressbooks.pub/rspnc/?p=209#h5p-80
- En utilisant les graphiques de base de R, créez un diagramme à moustaches comparant l’utilisation de l’espace occupée par les anoles verts par rapport aux anoles bruns. De nouveau, colorez le diagramme à moustaches de l’anole vert en vert, et celui de l’anole brun en brun.
An interactive H5P element has been excluded from this version of the text. You can view it online here:
https://ecampusontario.pressbooks.pub/rspnc/?p=209#h5p-81
- En utilisant ggplot, créez un nuage de points de l’utilisation de l’espace en fonction de l’agression moyenne. Ajoutez un facteur de couleur, de sorte que les points soient colorés en fonction de leur étiquette d’espèce (vert pour les anoles verts, brun pour les anoles bruns).
- Superposez une ligne de régression lm pour chaque espèce, ainsi qu’une mesure de l’erreur estimée pour ces lignes de régression.
- Positionnez votre légende de manière à ce qu’elle apparaisse dans un endroit vide pratique sur le panneau de la figure.
An interactive H5P element has been excluded from this version of the text. You can view it online here:
https://ecampusontario.pressbooks.pub/rspnc/?p=209#h5p-82
Fichiers à télécharger :
Pour télécharger, cliquez avec le bouton droit de la souris et appuyez sur “Enregistrer le fichier sous” ou “Télécharger le fichier lié”.
- invasivelizards.csv
Laboratoire et Institution ou Investigateur Principal :
Travail non publié – Candidat au doctorat Brendan McEwen
Références et lectures complémentaires :
Farrell, W. J., & Wilczynski, W. (2006). Aggressive experience alters place preference in green anole lizards, Anolis carolinensis. Animal Behaviour, 71(5), 1155-1164.
Rodríguez, C., Fusani, L., Raboisson, G., Hödl, W., Ringler, E., & Canoine, V. (2022). Androgen responsiveness to simulated territorial intrusions in Allobates femoralis males: evidence supporting the challenge hypothesis in a territorial frog. General and comparative endocrinology, 326, 114046.
C05: La socialité des mouches
Brendan McEwen
La socialité des mouches
Vous êtes un(e) sociobiologiste qui étudie la base génétique de la sociabilité chez les mouches à fruits. Une étude de dépistage génétique a identifié plusieurs “gènes candidats” qui pourraient jouer un rôle dans la régulation du comportement social chez Drosophila melanogaster. Vous décidez d’étudier le gène candidat Sec5, en réalisant une expérience de knockdown. Des cohortes de mouches mâles et femelles ont été soumises à l’interférence par ARN, éliminant ainsi l’activité du gène Sec5 chez ces individus. Une autre cohorte non modifiée génétiquement de mâles et de femelles a été conservée comme témoin. Vous avez rassemblé des groupes de mouches du même sexe dans des arènes de sociabilité, et vous avez suivi leur comportement d’agrégation dans le temps. Un “indice de sociabilité” a été calculé pour chaque groupe, des scores plus élevés indiquant que les mouches étaient plus étroitement regroupées les unes avec les autres – un signal plus fort de regroupement social.
Analysez cet ensemble de données pour voir si les mâles et les femelles diffèrent dans leurs niveaux de sociabilité, et si le silence du gène Sec5 a affecté la sociabilité chez les mâles ou les femelles
- Chargez les données et utilisez la commande head() pour prévisualiser le haut du dataframe.
An interactive H5P element has been excluded from this version of the text. You can view it online here:
https://ecampusontario.pressbooks.pub/rspnc/?p=211#h5p-83
- Créez une nouvelle colonne appelée condition, qui représente les combinaisons factorielles de sexe et de traitement génique.
An interactive H5P element has been excluded from this version of the text. You can view it online here:
https://ecampusontario.pressbooks.pub/rspnc/?p=211#h5p-84
- Dans GGplot, produisez un ensemble d’histogrammes d’indice de sociabilité pour les quatre combinaisons de conditions. Facettez le panneau par condition, de sorte que toutes les distributions des quatre conditions soient présentées sur le graphique en même temps.
An interactive H5P element has been excluded from this version of the text. You can view it online here:
https://ecampusontario.pressbooks.pub/rspnc/?p=211#h5p-85
- En utilisant la fonction lmer() du package lme4, créez un modèle linéaire mixte pour déterminer si le sexe, le traitement, ou leur interaction ont un effet significatif sur les scores d’indice de sociabilité sur les mouches dans votre expérience. Utilisez une approche de somme des carrés de type III pour analyser votre modèle, en utilisant la fonction Anova() du package car en R. Après avoir construit votre modèle, vérifiez ses diagnostics en utilisant la fonction check_model() du package performance
- Pour des informations sur la modélisation linéaire mixte, voir : Ce billet de blog
- Voir aussi : Cette vidéo instructive
- Indice : les variables ‘Arena’, ‘Time’, et ‘Day’ devraient être présentes dans vos effets aléatoires.
- Pour une introduction aux analyses ANOVA de type I / II / III, voir : Ce billet de blog
An interactive H5P element has been excluded from this version of the text. You can view it online here:
https://ecampusontario.pressbooks.pub/rspnc/?p=211#h5p-86
- En utilisant GGplot, produisez un graphique a pluie montrant les scores de sociabilité de chacune des quatre combinaisons de conditions.
An interactive H5P element has been excluded from this version of the text. You can view it online here:
https://ecampusontario.pressbooks.pub/rspnc/?p=211#h5p-87
Fichiers à télécharger :
Pour télécharger, cliquez avec le bouton droit de la souris et appuyez sur “Enregistrer le fichier sous” ou “Télécharger le fichier lié”.
- FlySociality.csv
Laboratoire et Institution ou Investigateur Principal :
Cognitive Ecology Lab, Dr. Reuven Dukas, Département de psychologie, neurosciences et comportement de l’Université McMaster https://psych.mcmaster.ca/dukas/index.htm
Références et lectures complémentaires :
Torabi-Marashi, A. (2023). Investigating the genetic basis of natural variation in sociability within Drosophila melanogaster (Doctoral dissertation)
Scott, A. M., Dworkin, I., & Dukas, R. (2022). Evolution of sociability by artificial selection. Evolution, 76(3), 541-553
C06 : Effets d’apprentissage dans les poissons subordonnés
Sina Zarini
Effets d’apprentissage dans les poissons subordonnés
En tant qu’écologiste comportemental à l’Institut de recherche Hamilton, vous vous plongez dans la dynamique sociale intéressante de Neolamprologus pulcher, le poisson cichlidé vivant en groupe originaire du lac Tanganyika en Afrique. Votre objectif principal est d’explorer l’impact du rang social sur les capacités d’apprentissage au sein des groupes de N. pulcher. Dans cette étude captivante, chaque unité sociale de N. pulcher comprend deux reproducteurs dominants et un nombre variable d’aides subordonnés, atteignant souvent jusqu’à 20 individus. Votre recherche se déroule à travers une série de trois expériences, toutes documentées dans un ensemble de données complet. L’ensemble de données englobe des variables clés, y compris l’ID du poisson, le sexe, la taille et trois aspects critiques de l’apprentissage :
Apprentissage initial (Expérience 1) : Les poissons ont été individuellement formés pour déplacer un disque coloré (bleu) pour découvrir une source de nourriture cachée. Les données sont dans la colonne “LearnedInitial”.
Apprentissage associatif (Expérience 2) : Un deuxième disque, de couleur jaune et immobile, a été introduit pour évaluer la capacité du poisson à l’associer à la nourriture. Les données sont dans la colonne “LearnedAssos”.
Apprentissage inversé (Expérience 3) : Dans cette expérience, la couleur du disque qui pouvait être déplacé a été changée, mettant au défi le poisson d’adapter et d’inverser leur comportement appris. Les données sont dans la colonne “LearnedReverse”.
1. Longueur du poisson dominant :
- Quelle est la longueur médiane du poisson dominant ?
- Quelle est la plage de longueurs pour le poisson dominant ?
- Quelle est la longueur moyenne du poisson dominant ?
- Veuillez créer un histogramme pour explorer visuellement la distribution des longueurs du poisson dominant.
An interactive H5P element has been excluded from this version of the text. You can view it online here:
https://ecampusontario.pressbooks.pub/rspnc/?p=213#h5p-88
2. Longueur du poisson subordonné :
- Quelle est la longueur médiane du poisson subordonné ?
- Quelle est la plage de longueurs pour le poisson subordonné ?
- Quelle est la longueur moyenne du poisson subordonné ?
- Générez un histogramme illustrant la distribution des longueurs du poisson subordonné.
An interactive H5P element has been excluded from this version of the text. You can view it online here:
https://ecampusontario.pressbooks.pub/rspnc/?p=213#h5p-89
3. Résultats d’apprentissage pour le poisson dominant :
- Quel pourcentage de poissons dominants ont réussi à apprendre la tâche initiale ?
- Quel pourcentage de poissons dominants ont réussi à associer le disque jaune à la nourriture ?
- Quel pourcentage de poissons dominants ont réussi à inverser leur apprentissage ?
- Créez des graphiques à barres pour représenter visuellement les pourcentages de poissons dominants qui ont réussi à apprendre la tâche initiale, à associer le disque jaune à la nourriture et à inverser leur apprentissage. Utilisez des nuances de rouge pour plus de clarté.
An interactive H5P element has been excluded from this version of the text. You can view it online here:
https://ecampusontario.pressbooks.pub/rspnc/?p=213#h5p-90
4. Résultats d’apprentissage pour le poisson subordonné :
- Quel pourcentage de poissons subordonnés ont réussi à apprendre la tâche initiale ?
- Quel pourcentage de poissons subordonnés ont réussi à associer le disque jaune à la nourriture ?
- Quel pourcentage de poissons subordonnés ont réussi à inverser leur apprentissage ?
- Générez des graphiques à barres indiquant les pourcentages de poissons subordonnés atteignant des résultats réussis dans les tâches d’apprentissage. Visualisez les données en utilisant des nuances de bleu.
An interactive H5P element has been excluded from this version of the text. You can view it online here:
https://ecampusontario.pressbooks.pub/rspnc/?p=213#h5p-91
5. Différences de taille :
- La distribution des longueurs de poissons est-elle normale ? (Effectuez un test de normalité)
- En fonction des résultats de normalité, quel test statistique serait approprié pour examiner les différences significatives dans les longueurs de poissons entre les dominants et les subordonnés ?
- Exécutez le test et rapportez le résultat. Veuillez fournir un diagramme à moustaches pour comparer les distributions de longueurs des poissons dominants et subordonnés.
An interactive H5P element has been excluded from this version of the text. You can view it online here:
https://ecampusontario.pressbooks.pub/rspnc/?p=213#h5p-92
Fichiers à télécharger :
- b06_simulated_learningdataset.csv
C07 : L’alimentation des poissons
Sina Zarini
L’alimentation des poissons
En tant que chercheur étudiant les habitudes alimentaires des gobies ronds juvéniles (Neogobius melanostomus) dans le port de Hamilton, vous vous plongez dans les subtilités de leur comportement alimentaire, de jour comme de nuit. Le goby rond, un petit poisson d’eau douce originaire d’Eurasie, s’est établi dans diverses régions d’Amérique du Nord, y compris les Grands Lacs. Ces gobies, connus pour leur appétit vorace et leur adaptabilité, jouent un rôle significatif dans les écosystèmes locaux en tant que prédateurs et proies.
Dans le port de Hamilton, les gobies ronds juvéniles se nourrissent principalement de deux types d’organismes : les cladocères et les rotifères. Votre ensemble de données offre des informations précieuses sur le contenu de l’estomac de ces juvéniles, révélant leurs habitudes de consommation à différents moments de la journée. Les variables enregistrées comprennent l’ID du poisson, l’heure de la collecte, et les quantités de cladocères et de rotifères trouvées dans l’estomac de chaque poisson.
1. Consommation de Cladocères et de Rotifères :
- Quel est le nombre médian de cladocères consommés par le goby rond juvénile ?
- Quelle est la plage du nombre de cladocères consommés ?
- Quel est le nombre moyen de rotifères consommés par le goby rond juvénile ?
- Générez des boîtes à moustaches pour explorer visuellement la distribution de la consommation de cladocères et de rotifères par le goby rond juvénile.
An interactive H5P element has been excluded from this version of the text. You can view it online here:
https://ecampusontario.pressbooks.pub/rspnc/?p=215#h5p-93
2. Modèles Alimentaires de Jour et de Nuit :
- Quel est le nombre médian de cladocères consommés pendant le jour par rapport à la nuit ?
- Quelle est la plage du nombre de rotifères consommés pendant le jour par rapport à la nuit ?
- Comparez le nombre moyen de cladocères et de rotifères consommés pendant le jour et la nuit.
- Créez des graphiques à boîtes séparées pour comparer visuellement la distribution de la consommation de cladocères et de rotifères pendant le jour et la nuit.
An interactive H5P element has been excluded from this version of the text. You can view it online here:
https://ecampusontario.pressbooks.pub/rspnc/?p=215#h5p-94
3. Préférence de Proie :
- Quel est le pourcentage de proies totales composées de cladocères ?
- Quel est le pourcentage de proies totales composées de rotifères ?
- Générez un graphique à barres empilées pour illustrer la composition des proies (cladocères et rotifères) dans le régime alimentaire du goby rond juvénile.
An interactive H5P element has been excluded from this version of the text. You can view it online here:
https://ecampusontario.pressbooks.pub/rspnc/?p=215#h5p-95
4. Comportement Alimentaire Global :
- Quel est le nombre total de proies consommées par le goby rond juvénile ?
- Y a-t-il une corrélation entre le nombre de cladocères et de rotifères consommés ?
- Tracez un graphique de dispersion pour visualiser la relation entre le nombre de cladocères et de rotifères consommés.
- Y a-t-il une différence significative entre la consommation de cladocères et de rotifères pendant le jour et la nuit (considérez les échantillons de jour et de nuit comme indépendants) ?
- Vérifiez d’abord la normalité, puis exécutez les tests appropriés.
An interactive H5P element has been excluded from this version of the text. You can view it online here:
https://ecampusontario.pressbooks.pub/rspnc/?p=215#h5p-96
Fichiers à télécharger :
- b07_simulated_diet.csv
C08 : Les comportements de dispersion
Sina Zarini
Les comportements de dispersion
Dans cette expérience, les chercheurs ont étudié le comportement de dispersion du gobie rond dans un environnement de laboratoire contrôlé, explorant comment il varie sous des conditions de jour et de nuit. De plus, les niveaux d’activité du gobie ont été enregistrés dans le réservoir expérimental. Chaque gobie a été catégorisé en fonction de sa taille (Petit, Moyen ou Grand) et s’il a montré un comportement de dispersion. Le niveau d’activité de chaque gobie a également été mesuré. L’ensemble de données comprend des informations cruciales sur la taille de chaque gobie, leur comportement de dispersion et leurs niveaux d’activité dans l’environnement expérimental.
1. Comportement de dispersion :
- Quel pourcentage de gobies ronds a montré un comportement de dispersion ?
- Parmi les gobies qui se sont dispersés, quel était le niveau d’activité moyen ?
- Créez un graphique à barres pour visualiser la proportion de gobies montrant un comportement de dispersion pour chaque catégorie de taille.
An interactive H5P element has been excluded from this version of the text. You can view it online here:
https://ecampusontario.pressbooks.pub/rspnc/?p=217#h5p-97
2. Niveaux d’activité :
- Quel était le niveau d’activité médian du gobie rond dans le réservoir expérimental ?
- Comparez les niveaux d’activité des gobies montrant un comportement de dispersion à ceux qui ne l’ont pas fait.
- Y a-t-il une différence significative ?
- Générez un graphique à boîtes pour comparer visuellement la distribution des niveaux d’activité entre les gobies qui ont montré un comportement de dispersion et ceux qui ne l’ont pas fait.
An interactive H5P element has been excluded from this version of the text. You can view it online here:
https://ecampusontario.pressbooks.pub/rspnc/?p=217#h5p-98
3. Taille et dispersion :
- Y a-t-il une association entre la taille du gobie rond et leur probabilité de dispersion ?
- Effectuez un test du chi-carré pour déterminer la signification.
- Calculez le pourcentage de gobies de chaque catégorie de taille qui ont montré un comportement de dispersion.
- Créez un graphique à barres empilées pour illustrer la distribution du comportement de dispersion parmi les gobies de différentes tailles.
An interactive H5P element has been excluded from this version of the text. You can view it online here:
https://ecampusontario.pressbooks.pub/rspnc/?p=217#h5p-99
4. Taille et activité :
- Y a-t-il une différence significative dans les niveaux d’activité parmi les différentes tailles de gobies ronds ?
- Visualisez la relation entre la taille du gobie et leurs niveaux d’activité à l’aide d’un graphique à boîtes, et effectuez une ANOVA à un facteur pour tester les différences.
An interactive H5P element has been excluded from this version of the text. You can view it online here:
https://ecampusontario.pressbooks.pub/rspnc/?p=217#h5p-100
Fichiers à télécharger :
- b08_simulated_dispersal.csv
C09: La dynamique des populations
Sina Zarini
La dynamique des populations
Enquêtant sur la dynamique de la population du gobie rond (Neogobius melanostomus) dans le lac Ontario, nous nous penchons sur les caractéristiques biologiques des individus échantillonnés dans ce lac. L’ensemble de données comprend des détails tels que le sexe, la longueur et la masse du poisson, essentiels pour comprendre la démographie de cette espèce envahissante. En analysant ces attributs, nous visons à obtenir des informations sur la distribution, les modèles de croissance et les impacts potentiels du gobie rond dans l’écosystème du lac.
1. Statistiques descriptives :
- Quelle est la longueur médiane du gobie rond échantillonné dans le lac Ontario ?
- Quelle est la plage de longueurs observée dans la population de gobies ronds échantillonnée ?
- Calculez la masse moyenne des spécimens de gobie rond.
- Calculez l’écart type des longueurs observées dans la population de gobies ronds échantillonnée.
- Calculez le pourcentage de gobies ronds mâles et femelles dans la population échantillonnée.
An interactive H5P element has been excluded from this version of the text. You can view it online here:
https://ecampusontario.pressbooks.pub/rspnc/?p=219#h5p-101
2. Analyse graphique :
Distribution de la longueur :
- Créez un histogramme pour visualiser la distribution des longueurs parmi les spécimens de gobie rond.
Distribution de la masse :
- Générez un histogramme pour visualiser la distribution des masses parmi les spécimens de gobie rond.
Relation longueur vs masse :
- Créez un nuage de points pour visualiser la relation entre la longueur et la masse parmi les spécimens de gobie rond.
Comparaison par sexe :
- Générez des boîtes à moustaches côte à côte pour comparer les distributions de longueurs et de masses entre les gobies ronds mâles et femelles.
An interactive H5P element has been excluded from this version of the text. You can view it online here:
https://ecampusontario.pressbooks.pub/rspnc/?p=219#h5p-102
3. Tests statistiques :
Analyse de corrélation :
- Vérifiez la normalité de la masse et de la longueur.
- Y a-t-il une corrélation entre la longueur et la masse parmi les spécimens de gobie rond ? Effectuez le test de corrélation approprié et rapportez le coefficient de corrélation.
Différences de sexe :
- Y a-t-il des différences significatives de longueur entre les gobies ronds mâles et femelles ?
Test d’hypothèse sur le ratio des sexes :
- Effectuez un test du chi-carré pour déterminer si le ratio des sexes observé dans la population de gobies ronds échantillonnée diffère significativement d’un ratio attendu de 1:1.
An interactive H5P element has been excluded from this version of the text. You can view it online here:
https://ecampusontario.pressbooks.pub/rspnc/?p=219#h5p-103
Fichiers à télécharger :
- b09_simulated_population.csv
C10: Les comportements de la natation
Sina Zarini
Les comportements de la natation
Dans notre étude, nous étudions le comportement de nage des gobies ronds juvéniles (Neogobius melanostomus) à l’aide d’une expérience de tunnel de nage. Cet environnement contrôlé nous permet d’observer deux comportements principaux : la nage active, caractérisée par un mouvement et une exploration continus, et le maintien en station, où les poissons maintiennent une position relativement stationnaire. En enregistrant ces comportements, nous visons à comprendre les modèles d’activité et les préférences du gobie rond en réponse à des conditions variables. Notre ensemble de données comprend des mesures des durées de nage et de maintien, ainsi que la catégorisation de la taille des poissons, fournissant des informations sur leur dynamique locomotrice et leurs interactions écologiques.
1. Statistiques descriptives :
- Quelle est la durée médiane de nage du gobie rond juvénile dans l’expérience de tunnel de nage ?
- Quelle est la plage des durées de nage observées parmi les spécimens de gobie rond juvénile ?
- Calculez la durée moyenne de nage du gobie rond juvénile dans l’expérience de tunnel de nage.
- Calculez l’écart type des durées de nage observées parmi les spécimens de gobie rond juvénile.
- Calculez le pourcentage de poissons catégorisés comme petits, moyens et grands dans la population échantillonnée.
An interactive H5P element has been excluded from this version of the text. You can view it online here:
https://ecampusontario.pressbooks.pub/rspnc/?p=221#h5p-104
2. Analyse graphique :
- Créez un histogramme pour visualiser la distribution des durées de nage parmi les spécimens de gobie rond juvénile.
- Générez un histogramme pour visualiser la distribution des durées de maintien parmi les spécimens de gobie rond juvénile.
Relation Durée de Nage vs Durée de Maintien :
- Créez un nuage de points pour visualiser la relation entre les durées de nage et de maintien parmi les spécimens de gobie rond juvénile.
Comparaison par Taille :
- Générez des boîtes à moustaches côte à côte pour comparer les distributions des durées de nage et de maintien entre les gobies ronds petits, moyens et grands.
An interactive H5P element has been excluded from this version of the text. You can view it online here:
https://ecampusontario.pressbooks.pub/rspnc/?p=221#h5p-105
3. Tests statistiques :
- Vérifiez la normalité des durées de nage et de maintien à l’aide d’un graphique Q-Q et du test de Shapiro-Wilk.
- Y a-t-il une corrélation entre les durées de nage et de maintien parmi les spécimens de gobie rond juvénile ? Effectuez un test de corrélation et rapportez le coefficient de corrélation.
- Y a-t-il des différences significatives dans les durées de nage entre différentes catégories de taille de gobie rond juvénile ? Effectuez le test approprié et interprétez les résultats. Effectuez des tests post-hoc si nécessaire.
An interactive H5P element has been excluded from this version of the text. You can view it online here:
https://ecampusontario.pressbooks.pub/rspnc/?p=221#h5p-106
Fichiers à télécharger :
- b10_simulated_swimming.csv















 `eegtime` Creates plot of single-channel electroencephalography (EEG) time course with optional confidence
interval. User can control the plot orientation, line types, line colors, etc.
`eegtime` Creates plot of single-channel electroencephalography (EEG) time course with optional confidence
interval. User can control the plot orientation, line types, line colors, etc.